QC(质控)-HBC lesson 4
在这一章你将会学到:
- 构建质控指标并使用相关图像可视化数据质量
- 估算质控指标并设置阈值去除低质量的细胞
单细胞数据分析流程的每个步骤都有这自己的目标和挑战。对于我们原始计数数据 (raw count data) 的质量控制,它们包括:
目的:
- 为了过滤数据,使其只包含高质量的真实细胞,这样当我们对细胞进行聚类时,就更容易识别不同的细胞类型群体
- 识别出任何低质量的样本,尝试修正或从分析中删除,此外,尝试了解样本质量较低的原因
面临的挑战:
- 从复杂性低的细胞中分离出低质量的细胞
- 选择合适的过滤阈值 (threshold),以保留高质量的细胞而不去除与生物学相关的细胞类型。
建议:
在进行质量控制之前,首先要对自己样本中可能存在的细胞类型有一定的预期。例如,你的样本中是不是有可能复杂程度较低或者其中可能含有线粒体表达水平较高的细胞类型?如果是这样,那么在评估数据质量时,我们需要考虑到这种生物学特征。
生成质量指标 (Generating quality metrics)
请记住,Seurat会自动为每个细胞创建一些元数据:
# 查看合并的元数据
View(merged_seurat@meta.data)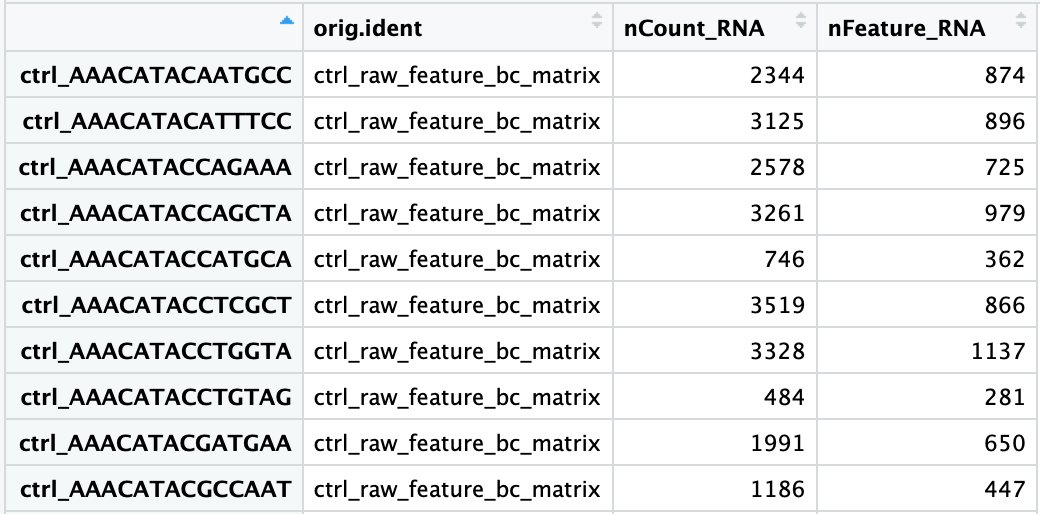
这些添加的列包括:
orig.ident:通常包含所知的样品名,默认为我们赋给project的值nCount_RNA:每个细胞的UMI数目nFeature_RNA:每个细胞所检测到的基因数目
我们需要额外计算添加一些可用于可视化的其他指标:
每个UMI检测到的基因数目 (number of genes detected per UMI)
这个指标让我们了解数据集的复杂性(每个UMI检测到的基因越多,我们的数据越复杂)
每个细胞的每个UMI的基因数很容易计算,我们取log10转换结果,以便更好地进行样本之间的比较。
# 将每个细胞每个UMI的基因数目添加到元数据中
merged_seurat$log10GenesPerUMI <- log10(merged_seurat$nFeature_RNA) / log10(merged_seurat$nCount_RNA)线粒体比率 (mitochondrial ratio)
这个指标将给我们一个来自于线粒体基因的细胞读取的占比
Seurat有一个方便的功能,可以让我们计算转录本映射到线粒体基因的比例 PercentageFeatureSet() 将使用一个模式(pattern)搜索基因标识符。对于每一列(细胞),它将选取特征基因的计数之和,除以所有基因的计数之和,再乘以100。因为我们想要绘制占比值 (ratio value),所以我们可以通过除以100把这个步骤颠倒过来。
# 计算线粒体比率
merged_seurat$mitoRatio <- PercentageFeatureSet(object = merged_seurat, pattern = "^MT-")
merged_seurat$mitoRatio <- merged_seurat@meta.data$mitoRatio / 100注:提供的模式 (pattern) ("^MT") 适用于人类基因名。你可能需要根据你感兴趣的物种进行调整。如果不使用基因名作为基因ID,那么这个函数就不起作用。更准确的方法可以使用额外的注释文件,进行计算。
将元数据信息提取为单独变量
我们需要为质控指标在元数据中添加额外的信息,比如细胞ID、条件信息和其他各种指标。虽然使用$操作符将信息直接添加到Seurat对象的元数据槽中非常容易,但是我们将把数据框提取到一个单独的变量中。这样,我们可以继续插入我们的质控分析所需的额外指标,而不会影响merged_seurat对象。
我们将从Seurat对象中提取meta.data插槽来构建元数据数据框:
# 将元数据提取为单独变量(这一步也可以不做,直接操作元数据也可以)
metadata <- merged_seurat@meta.data你应该看到每个细胞ID都有一个ctrl_或stim_前缀,正如我们在合并Seurat对象时指定的那样。这些前缀应符合orig.ident中列出的样本。让我们首先添加一个具有细胞ID的列,并更改当前的列名以使其更直观:
# 为元数据添加细胞ID
metadata$cells <- rownames(metadata)
# 重命名列
metadata <- metadata %>%
dplyr::rename(seq_folder = orig.ident,
nUMI = nCount_RNA,
nGene = nFeature_RNA)现在,让我们根据细胞前缀获取每个细胞的样本名称:
# 创建样本列
metadata$sample <- NA
metadata$sample[which(str_detect(metadata$cells, "^ctrl_"))] <- "ctrl"
metadata$sample[which(str_detect(metadata$cells, "^stim_"))] <- "stim"现在,你已经准备好了评估数据质量所需的全部指标!最终的元数据表将包含对应于每个细胞的行,以及包含有关这些细胞信息的列:

将更新的元数据保存至我们的Seurat对象
在我们评估指标标准之前,我们将把迄今为止所做的所有工作都保存到Seurat对象中。我们可以通过简单地将数据框赋值到meta.data位置中来实现:
# 将元数据添加回Seurat对象中
merged_seurat@meta.data <- metadata
# 任何时候都要创建.RData对象保存进度
save(merged_seurat, file="data/merged_filtered_seurat.RData")评估质量指标
现在我们已经生成了要评估的各种指标,我们可以用可视化的方式来探索它们。我们将评估各种指标,然后决定哪些细胞质量较低,而应从分析中删除:
- 细胞计数 (Cell counts)
- 每个细胞的UMI计数 (UMI counts per cell)
- 每个细胞检测到的基因 (Genes detected per cell)
- 检测到的UMI数对比基因数 (UMIs vs. genes detected)
- 线粒体计数比率 (Mitochondrial counts ratio)
- 复杂度 (Novelty)
那么双胞 (doublet)为什么不在其中 ?
在单细胞RNA测序实验中,双胞是由两个细胞产生的。它们通常是由于细胞分选或捕获错误而产生,特别是在涉及数千个细胞的基于液滴的流程中。当目的是为了在单细胞水平上区分种群时,双胞显然是不可取的。尤其是,他们可能错误暗示实际上并不存在的中间类群或过渡状态。因此,最好删除双胞文库,以免影响对结果的诠释。为什么我们不检查双胞?许多工作流程使用最大阈值来检测UMI或基因,其想法是检测到的读取或基因数量异常多的细胞表示多重细胞。虽然这个道理似乎很直观,但并不准确。此外,许多用于检测双胞的工具往往会去掉具有中间或连续表型的细胞,尽管它们可能在离散性强的细胞类型数据集上效果很好。Scrublet是一个流行的双胞检测工具,但我们还没有对其进行充分的基准测试。目前,我们建议在这个时候不包括任何阈值。可先进行分析,细胞分群,鉴定出相应的类群标记物之后,对标记物研究进一步作出判断(是双胞类群还是过渡状态细胞)。
细胞计数 (Cell counts)
细胞计数是由检测到的独特的细胞条码数量来确定。在这个实验中,预计会有12,000-13,000个细胞。
理想情况下,UMIs数量与你测序装载的细胞数量相关。然而,事实并非如此,因为捕获的细胞只是装载细胞数量的一部分。例如,inDrops的细胞捕获效率较高(70-80%),而10X的细胞捕获率大约在50-60%之间。
注:如果用于建库的细胞浓度不准确,则捕获效率可能会显得低得多。细胞浓度不应该由FACS机器或Bioanalyzer确定(这些工具的浓度测定是不准确的),而应使用血细胞仪或自动细胞计数器计算细胞浓度。
细胞数也可以因方法而异,而产生比我们装载的高得多的细胞数。例如,在inDrops方法中,细胞条码存在于水凝胶中,同单个细胞和裂解/反应混合物一起被封装在液滴内。虽然每个水凝胶应该有一个单一的细胞条码与之相关联,但偶尔一个水凝胶可以有一个以上的细胞条码。同样,在10X方法这里,有一定的几率仅获得在乳化液滴(GEM)中的条码珠子,而没有实际的细胞。这两点,除了濒死细胞的存在之外,还可能导致比细胞数更多的细胞条码。
# 可视化每个样本的细胞计数
metadata %>%
ggplot(aes(x=sample, fill=sample)) +
geom_bar() +
theme_classic() +
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust=1)) +
theme(plot.title = element_text(hjust=0.5, face="bold")) +
ggtitle("NCells")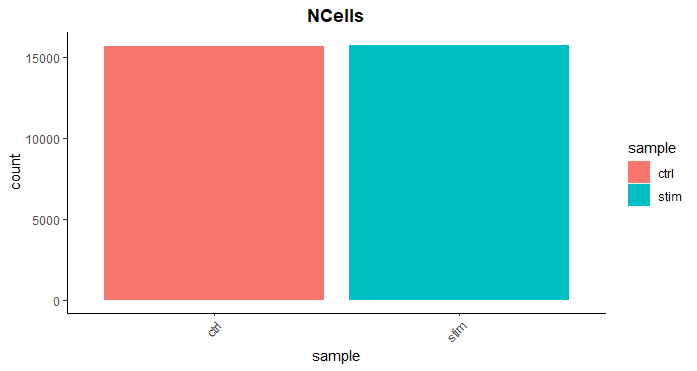
我们看到每个样本有超过15,000个细胞,这比预期的12-13,000个多了不少。很显然,我们很可能有一些垃圾 "细胞 "存在。
每个细胞的UMI计数 (UMI counts per cell)
每个细胞的UMI计数一般应该在500以上,这是我们预期的底线。如果UMI计数在500-1000个计数之间,也是可用的,但细胞可能应该进行更深度的测序。
# 可视化每个细胞的UMI/转录本数目
metadata %>%
ggplot(aes(color=sample, x=nUMI, fill= sample)) +
geom_density(alpha = 0.2) +
scale_x_log10() +
theme_classic() +
ylab("Cell density") +
geom_vline(xintercept = 500)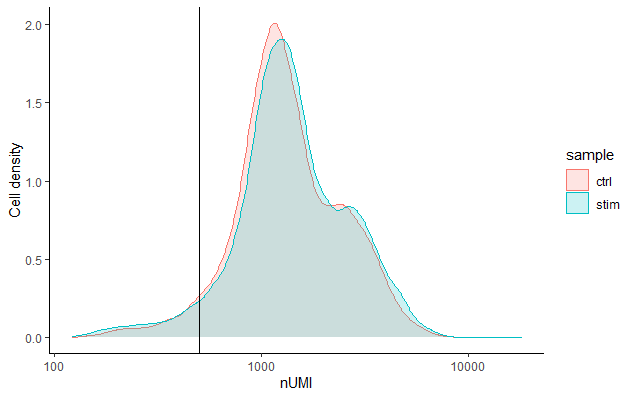
我们可以看到,在这两个样本中,我们大部分的细胞都有1000个或以上的UMI,这是很好的。
每个细胞检测到的基因 (Genes detected per cell)
我们对基因检测的期望值与UMI检测相似,尽管它可能比UMI低一些。对于高质量的数据,比例直方图应该存在一个单峰,将捕获的细胞囊括。如果我们看到大峰左侧有一个小的肩部(在我们的数据中没有出现),或者细胞的双态分布,这可能说明出了一点问题。这表示可能存在一些因为某种原因而建库失败的细胞。这也可能是存在生物学上不同类型的细胞(如静止的细胞群,复杂性低的细胞),和/或一种比其他类型小得多的细胞(比如高计数的细胞可能是体积较大的细胞)。因此,这个阈值应与我们在本文中描述的其他指标一起评估。
# 通过频数图可视化每个细胞检测出的基因数分布
metadata %>%
ggplot(aes(color=sample, x=nGene, fill= sample)) +
geom_density(alpha = 0.2) +
theme_classic() +
scale_x_log10() +
geom_vline(xintercept = 300)
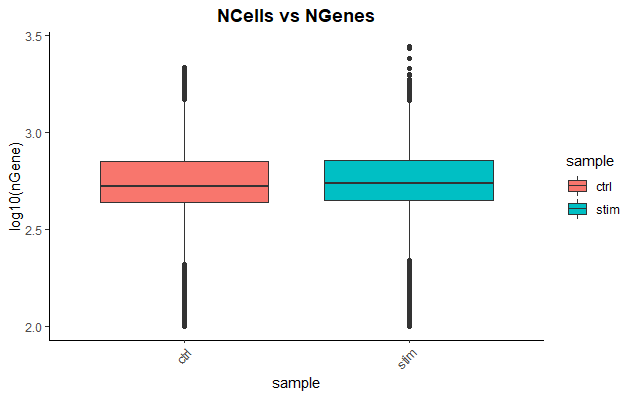
# 通过箱线图可视化每个细胞检测到的基因的分布
metadata %>%
ggplot(aes(x=sample, y=log10(nGene), fill=sample)) +
geom_boxplot() +
theme_classic() +
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust=1)) +
theme(plot.title = element_text(hjust=0.5, face="bold")) +
ggtitle("NCells vs NGenes")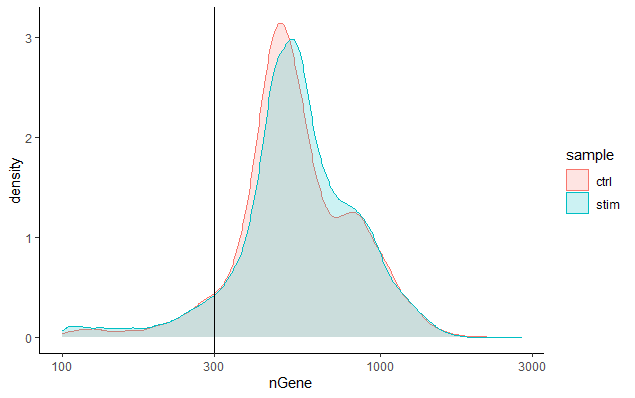
检测到的UMI数对比基因数 (UMIs vs. genes detected)
经常一起评估的两个指标是UMI的数量和每个细胞检测到的基因数量。在这里,我们已经绘制了基因数量对比UMI数量的散点图(线粒体序列占比着色)。线粒体序列占比只有在检测到很少基因的低计数细胞中是高的(深色)。这可能表明受损/死亡细胞的细胞质mRNA已经通过破膜漏出,因此,只有位于线粒体中的mRNA仍然保留。这些细胞被我们的计数和基因数阈 值过滤掉。将计数阈值和基因数量阈值联合可视化,显示了综合过滤效果 (joint filtering effect)。
质量差的细胞很可能具有较低的基因数和UMI数,并对应于该图左下象限的数据点。好的细胞一般基因数和UMI的数量都较高。
通过该图,我们还评估了该线的斜率,以及该图右下角象限中的散点数据。这些细胞有较高的UMI的数量,但只有少数基因的数量。这些可能是濒死的细胞,但也可能代表低复杂性细胞类型(即红细胞)的群体。
# 可视化检测到的基因数和UMI数之间的关系,并且观察是否存在大量低数目的基因数/UMI数的细胞
metadata %>%
ggplot(aes(x=nUMI, y=nGene, color=mitoRatio)) +
geom_point() +
scale_colour_gradient(low = "gray90", high = "black") +
stat_smooth(method=lm) +
scale_x_log10() +
scale_y_log10() +
theme_classic() +
geom_vline(xintercept = 500) +
geom_hline(yintercept = 250) +
facet_wrap(~sample)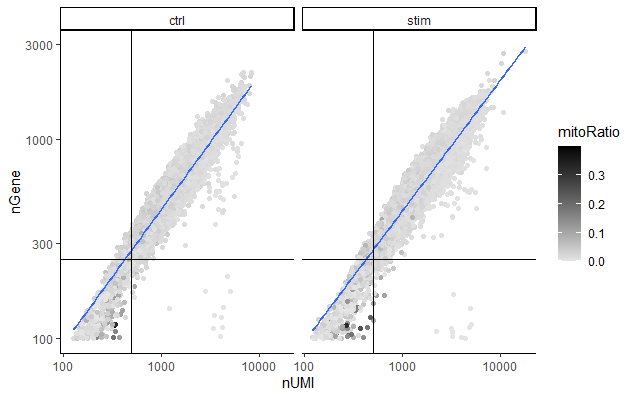
线粒体计数占比 (Mitochondrial counts ratio)
这个指标可以确定是否有大量死亡或濒死细胞的线粒体污染。我们通常把线粒体占比超过0.2的细胞定义为线粒体计数质量差的样品。(实际上也需要根据样本的具体情况进行调整,如心脑肺等组织中代谢旺盛,线粒体基因序列可能更高)
# 可视化每个细胞检测到的线粒体基因表达分布
metadata %>%
ggplot(aes(color=sample, x=mitoRatio, fill=sample)) +
geom_density(alpha = 0.2) +
scale_x_log10() +
theme_classic() +
geom_vline(xintercept = 0.2)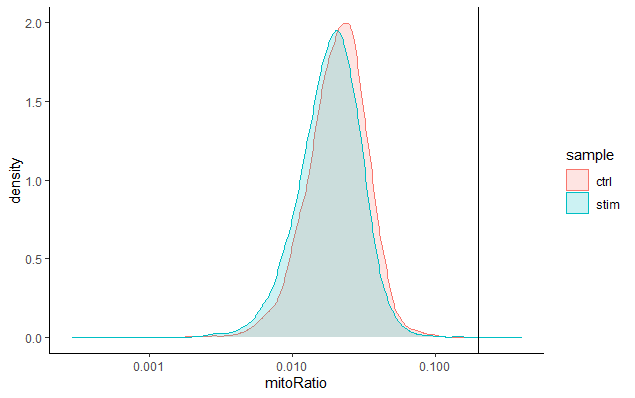
复杂度 (Complexity) ?
我们可以看到我们对每个细胞测序较浅的样本有较高的整体复杂性,这是因为这些样本中测到的基因都没有达到饱和测序。这些样本中的离群细胞可能是具有比其他细胞复杂度更低的RNA种类的细胞。有时,我们可以通过这个指标检测到低复杂度细胞类型的污染,比如红细胞等低复杂度细胞。一般情况下,我们期望复杂度得分在0.80以上。
# 通过可视化每一个UMI检测到的基因数来可视化基因表达的整体复杂性
metadata %>%
ggplot(aes(x=log10GenesPerUMI, color = sample, fill=sample)) +
geom_density(alpha = 0.2) +
theme_classic() +
geom_vline(xintercept = 0.8)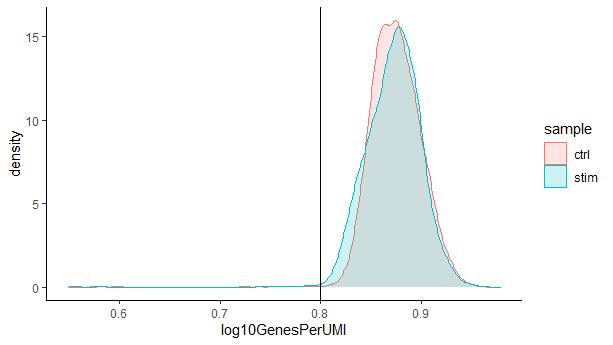
注:每个细胞的读取 (Reads per cell) 是另一个可以研究的有用指标;然而,所使用的工作流程需要保存这个信息来评估。一般来说,对于这个指标,你希望看到所有的样品与峰在相对相同的位置,每个细胞的读取在10,000到100,000之间
筛选 (Filtering)
孤立地考虑这些质控指标中的任何一个,都可能导致对细胞信息的误读。例如,线粒体计数比例相对较高的细胞可能参与呼吸过程,可能是你想保留的细胞。同样,其他指标也可能有其生物学上的解释。因此,在设置阈值时,总是要考虑到这些指标的共同影响,并尽可能地将其设置为允许的,以避免无意中过滤掉可用的细胞群。
细胞水平筛选 (Cell-level filtering)
现在,我们已经将各种指标可视化了,我们可以决定应用哪些阈值来去除低质量的细胞。通常情况下,前面提到的建议只是一个粗略的指导原则,具体的实验需要参考具体的阈值选择。我们将使用以下阈值。
- nUMI > 500
- nGene > 250
- log10GenesPerUMI > 0.8
- mitoRatio < 0.2
为了过滤,我们将回到Seurat对象,使用subset()函数。
# 使用选择的阈值筛掉低质量读写 - 这些将会随实验而改变
filtered_seurat <- subset(x = merged_seurat,
subset= (nUMI >= 500) &
(nGene >= 250) &
(log10GenesPerUMI > 0.80) &
(mitoRatio < 0.20))对于nUMI和nGene而言,一般只设置下限,如果设置上限建议尽量纳入较多的细胞,尽管高UMI或nGene的barcode可能是双细胞或多细胞,但是这些双细胞在下游分群的时候会显现出来,到时候再去除也不迟。
基因水平筛选 (Gene-level filtering)
在我们的数据中,会有许多基因的计数为零。这些基因会显著降低一个细胞的平均表达量,因此我们将从数据中删除它们。首先,我们将删除所有细胞中零表达的基因。此外,我们将按表达率进行一些筛选。如果一个基因只在少数几个细胞中表达,那么它的意义并不是特别大,因为它仍然会把没有表达它的其他所有细胞的平均表达量降下来。对于我们的数据,我们选择只保留在10个或更多细胞中表达的基因。
# 教程使用3.0版本,使用seurat v4.0 此方法不用学,但是可以看一下操作思路
# 提取计数
counts <- GetAssayData(object = filtered_seurat, slot = "counts")
# 根据在每个细胞的计数是否大于0为每个基因输出一个逻辑向量
nonzero <- counts > 0
# 将所有TRUE值相加,如果每个基因的TRUE值超过10个,则返回TRUE。
keep_genes <- Matrix::rowSums(nonzero) >= 10
# 仅保留那些在10个以上细胞中表达的基因
filtered_counts <- counts[keep_genes, ]
# 重新赋值给经过过滤的Seurat对象
filtered_seurat <- CreateSeuratObject(filtered_counts, meta.data = filtered_seurat@meta.data)这一步在seurat v4.0中可以直接在 CreateSeuratObject()函数中添加参数min.cells = 10即可,非常方便。但是如果创建对象的时候没有设置,后面需要调整可以用该方法。并且在首次创建seurat对象时设置的min.cell参数是对于单个样本而言的,后续需要对合并后的数据进一步处理可以使用此方法。
重新评估质量指标 (Re-assess QC metrics)
在进行过滤后,建议回过头来再看看指标,确保你的数据符合你的预期,对下游分析有好处。
保存筛选过的细胞 (Saving filtered cells)
根据这些质量控制指标,我们将识别出任何不合格的样本,然后继续进行筛选。通常情况下,我们会使用不同的过滤标准对质量控制指标进行迭代,这不一定是一个线性的过程。当对筛选标准满意后,我们将保存筛选后的细胞对象用于聚类 (clustering) 和标记识别 (marker identification)。
# 创建.RData对象以方便在任何时候导入
save(filtered_seurat, file="data/seurat_filtered.RData")原文来自:Harvard Chan Bioinformatics Core (HBC)
补充
在seurat v4.0中可以直接调用seurat自带的函数可视化QC参数(其实本质是也是在调用ggplot2包,只是整合到seurat中使用起来更方便,详见 Seurat基本分析流程
比如,我们把上述步骤筛选过的数据拿出来绘制小提琴图看一下:
VlnPlot(filtered_seurat, features = c("nUMI", "nGene", "mitoRatio"), ncol = 3, group.by = "sample")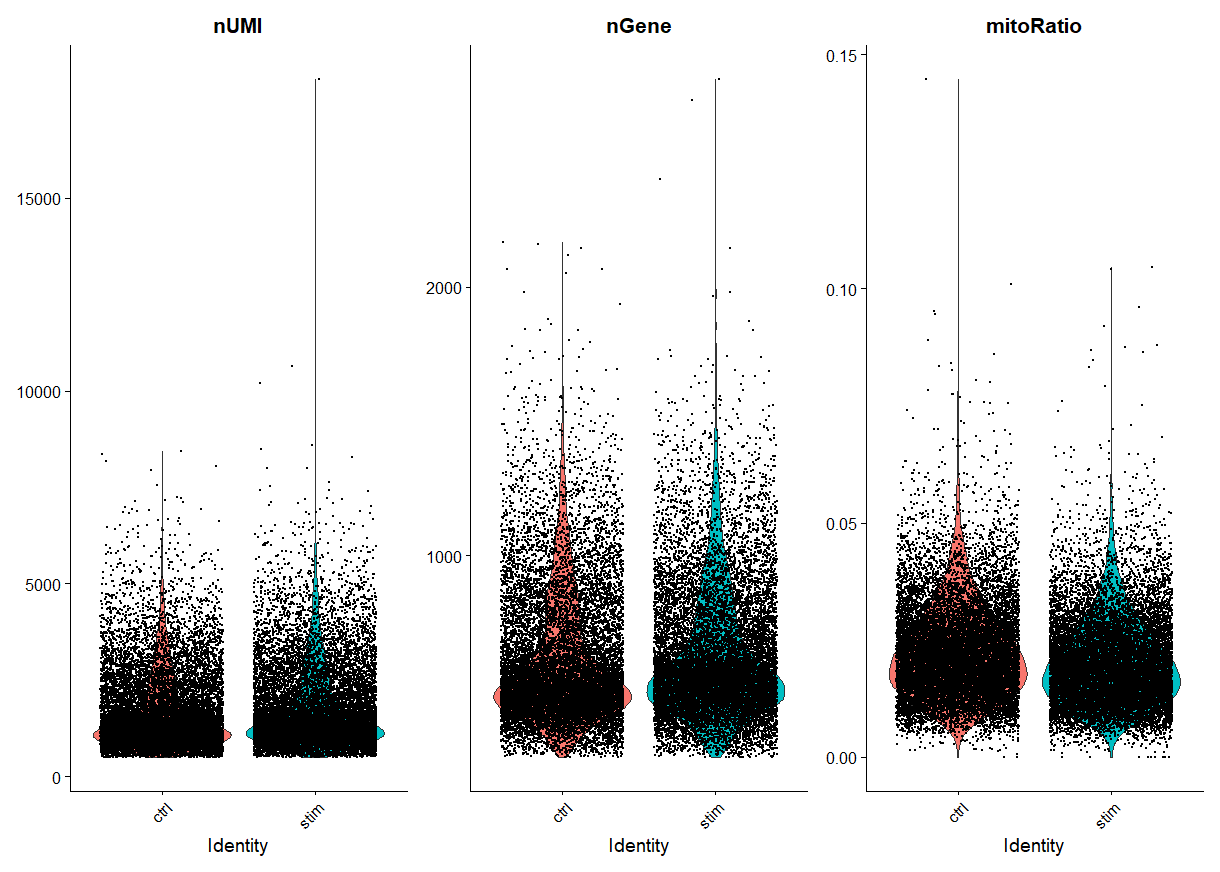
绘制散点图:
plot1 <- FeatureScatter(filtered_seurat, feature1 = "nUMI", feature2 = "mitoRatio", group.by = "sample")
plot2 <- FeatureScatter(filtered_seurat, feature1 = "nUMI", feature2 = "nGene", group.by = "sample")
plot1 + plot2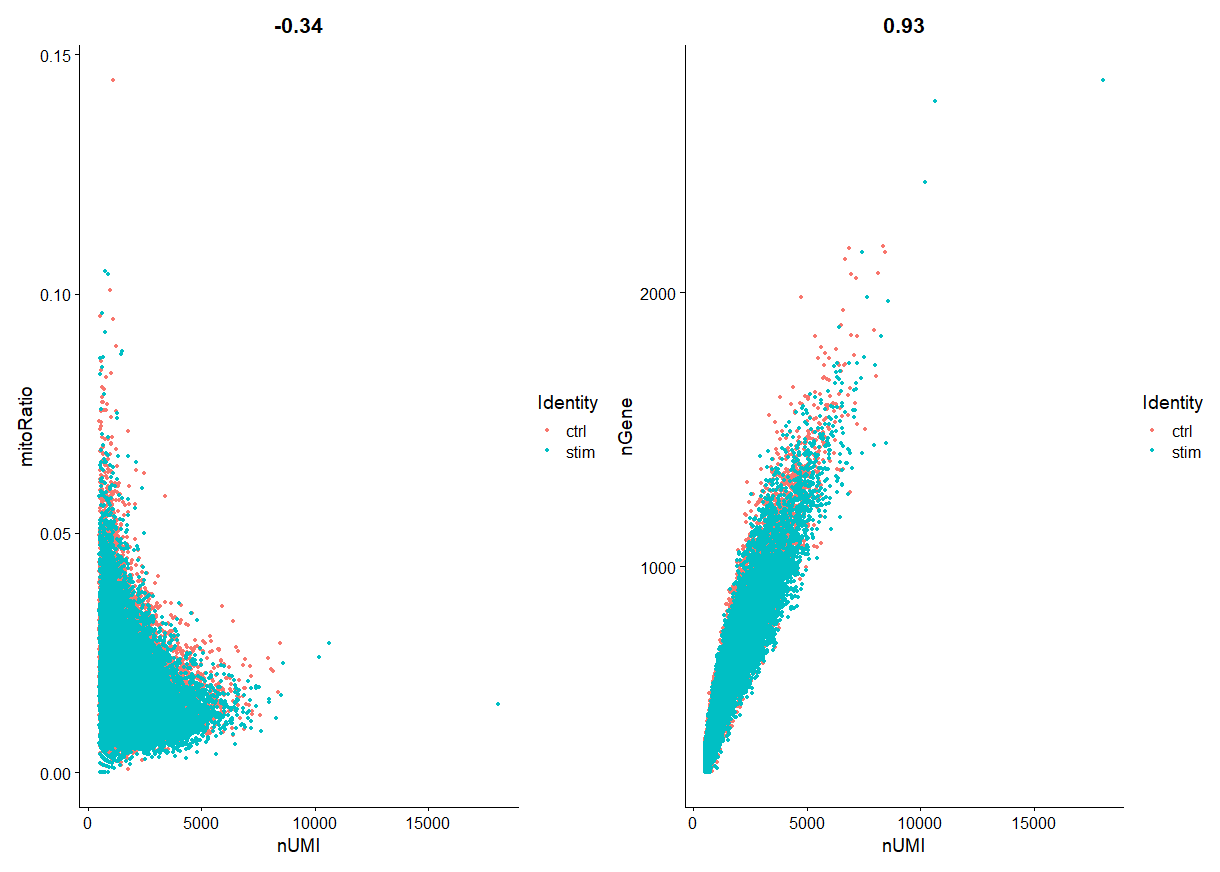











暂无评论内容