目标:
- 确定每个类群的基因标记物
- 使用标记物识别每个类群的细胞类型
- 根据细胞类型标记物来判断是否需要重新分组,或许需要合并或拆分聚类的类群
挑战:
- 对结果的过度解读
- 结合不同类型的标记物识别
建议:
- 将结果视为需要验证的假设。过高的p值会导致对结果的过度解读(基本上每个细胞都被当作一次重复)。排名靠前的标记物是最值得信赖的。识别在每个类群在任何处理条件下都保守的所有标记物。
- 识别出特定类群之间有不同表达的标记物。
我们的聚类分析结果如下:
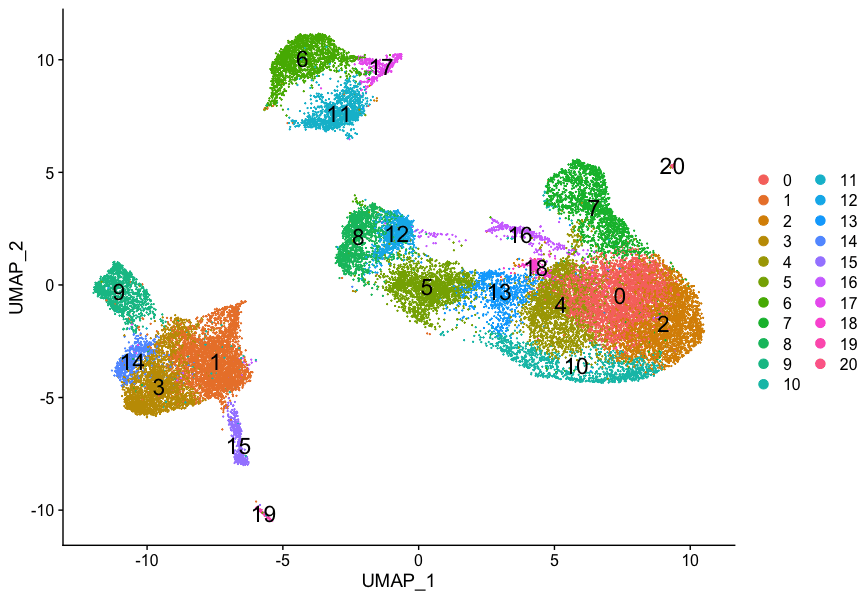
记得我们在聚类分析中,有以下几个问题:
- 类群7和类群20的细胞类型特征是什么?
- 与相同细胞类型对应的类群是否有生物学意义上的差异?这些细胞类型是否存在亚群?
- 我们是否可以通过识别这些细胞群的其他标记基因来获得更高的置信度?
有几种不同类型的标记基因鉴定,我们可以通过使用Seurat来探索这些问题的答案。每种类型都有各自的优点和缺点:
- 识别每个类群的所有标记物:这种分析将每个类群与其他所有类群进行比较,并输出不同表达/存在的基因。
- 有利于识别未知类群,提高对假设细胞类型的置信度。
- 识别每个类群的保守标记:该分析首先寻找在每个条件下不同表达/存在的基因,然后找出在所有条件下的类群中保守的基因。这些基因可以帮助找出该类群的特性。
- 对一个以上的条件下的细胞类型标记物识别非常有用,可以识别出跨条件下保守的细胞类型标记物。
- 特定类群之间的标记识别:该分析探索特定类群之间的不同表达基因。
- 有助于从上述分析中确定代表同一细胞类型(比如具有相似的标记物)的聚类之间的基因表达差异。
识别每个类群的全部标记物
这种类型的分析通常推荐用于评估单个样本组/条件。通过FindAllMarkers()函数,我们将每个类群与所有其他类群进行比较,以确定潜在的标记基因。每个类群中的细胞被视为重复,本质上是用一些统计检验进行差异表达分析。

注:默认为Wilcoxon Rank Sum检验,但也有其他选项。
FindAllMarkers()函数有三个重要的参数,这些参数提供了判断一个基因是否为标记物的阈值:
logfc.threshold:类群中基因的平均表达量相对于所有其他类群的平均表达量的最小log2倍数。默认值为0.25。- 缺点:
- 如果平均log2fc不符合阈值,可能会错过那些在感兴趣的聚类中小部分细胞表达,但在其他聚类中没有表达的细胞标记。
- 由于不同细胞类型的代谢输出略有差异,可能会返回大量代谢/核糖体基因,这对区分细胞类型的身份没有那么有用。
- 缺点:
min.diff.pct:该类群中表达该基因的细胞百分比与其他类群中表达该基因的细胞百分比之间的最小差异。- 缺点:可能漏掉那些在所有细胞中表达,但在该特定细胞类型中高度上调的细胞标记。
min.pct:只检验两个类群中任何一个类群中最少部分细胞中检测到的基因。旨在通过不检验那些表达频率很低的基因来加速函数运行。默认值为0.1。- 缺点:如果设置为一个很高的值,可能会产生很多假阴性,因为不是所有的基因都会在所有的细胞中检测到(即使是表达的基因)。
你可以使用这些参数的任意组合,这取决于你想有多严格/宽松。另外,默认情况下,这个函数会返回给你基因,这些基因同时表现出正向和负向表达的变化。通常情况下,我们添加一个参数only.pos来选择只保守表达上调的基因。为每个类群寻找标记的代码如下图所示。我们将不运行此代码。
## 不要运行次代码 ##
# 在所有剩余的细胞中找到每个类群的标记物,只报告上调的基因
markers <- FindAllMarkers(object = seurat_integrated,
only.pos = TRUE,
logfc.threshold = 0.25)识别所有条件下保守的标记物
由于我们数据集中的样本代表着不同的条件,所以我们最好的选择是寻找保守标记物 (conserved markers)。这个函数在类群内部按样本组/条件将细胞分离出来,然后对单个指定的类群与所有其他类群(或第二个类群,如果指定的话)进行差异基因表达检验。每个条件下基因水平的p值被计算出来,然后使用MetaDE R软件包中的元分析方法进行跨组合并。

在开始标记识别之前,我们将明确设置我们的默认分析方法,我们希望使用原始计数而不是聚合数据。
DefaultAssay(seurat_integrated) <- "RNA"注:虽然这个函数的默认设置是从 "RNA "插槽中获取数据,但我们建议你运行上面的这行代码,以防止在分析的上游某处改变了默认分析。原始计数和归一化计数都存储在这个位置中,用于查找标记的函数将自动提取原始计数。
函数FindConservedMarkers(),有以下结构:
FindConservedMarkers()语法:
FindConservedMarkers(seurat_integrated,
ident.1 = cluster,
grouping.var = "sample",
only.pos = TRUE,
min.diff.pct = 0.25,
min.pct = 0.25,
logfc.threshold = 0.25)你会认识到我们之前为FindAllMarkers()函数描述的一些参数;这是因为它在内部使用该函数时首先在每个组别中寻找标记。在这里,我们列出了一些额外的参数,这些参数是在使用FindConservedMarkers()函数时提供的。
ident.1:这个函数每次只评估一个类群;在这里你会指定感兴趣的类群。grouping.var:元数据中的变量(列头),它将会把细胞分组。
对于我们的分析,我们将相当宽松,只使用大于0.25的log2倍数变化作为阈值。我们还将指定只返回每个类群的上调标记。
让我们在一个类群上测试一下,看看它是如何工作的。
cluster0_conserved_markers <- FindConservedMarkers(seurat_integrated,
ident.1 = 0,
grouping.var = "sample",
only.pos = TRUE,
logfc.threshold = 0.25)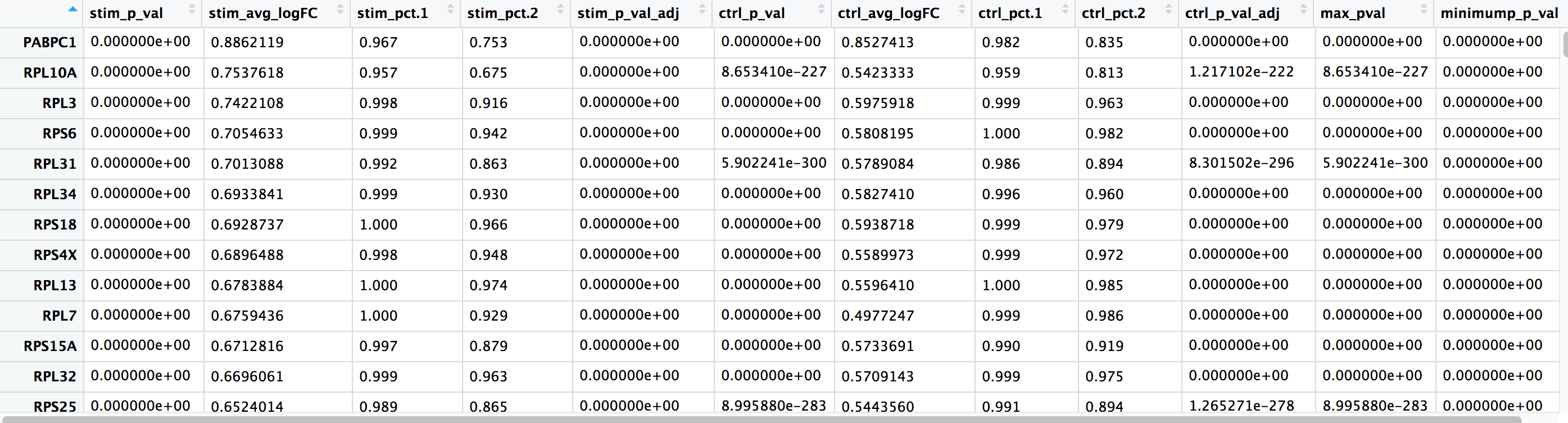
FindConservedMarkers()函数的输出,是一个矩阵,包含了一个按基因ID列出的假定标记物的排序列表,以及相关的统计数字。注意,每个组(在我们的例子中,Ctrl和Stim)都计算了同一组统计数字,最后两列对应的是两组之间的综合p值。我们将在下面描述其中的一些列名。
gene:基因符号condition_p_val: 未根据条件进行多重检验校正调整的p值condition_avg_logFC:条件的平均log2倍数变化。正值表示该基因在类群中表达较高。condition_pct.1:在条件下检测到该基因在类群中的细胞表达百分比。condition_pct.2:在条件的其他群组中平均检测到该基因的细胞表达百分比condition_p_val_adj:条件的调整后的p值,基于bonferroni校正,使用数据集中的所有基因,用于确定显著性max_pval: 各组/条件计算出的最大p值的p值。minimump_p_val: 合并的p值
注:由于每个细胞被当作一次重复处理,这将导致每个组内的p值被夸大!一个基因可能有一个非常低的p值<1e-50,但并不能转化为高度可靠的标记基因。
在看输出时,我们建议寻找pct.1和pct.2之间表达差异较大的标记基因,以及较大的倍数变化。例如,如果pct.1 = 0.90,pct.2 = 0.80,可能就不是那么需要注意的标记基因。然而,如果pct.2=0.1的话,差异更大,更有说服力。另外,如果大多数表达该标记的细胞在感兴趣的聚类中是比较有意义的。如果pct.1很低,比如0.3,可能就没有那么值得注意了。这两个参数也是如上所述运行函数时可能包含的参数。
添加基因注释 (Adding Gene Annotations)
添加带有基因注释信息的列会很有帮助。为了做到这一点,我们需要你将这个文件下载到你的data文件夹中,右击 "另存为…"。然后将其加载到你的R环境中。
annotations <- read.csv("data/annotation.csv")首先,我们将把带有基因标识符的行名变成它自己的列。然后我们将把这个注释文件与我们的FindConservedMarkers()结果合并。
# 将标记物和基因描述相结合
cluster0_ann_markers <- cluster0_conserved_markers %>%
rownames_to_column(var="gene") %>%
left_join(y = unique(annotations[, c("gene_name", "description")]),
by = c("gene" = "gene_name"))
View(cluster0_ann_markers)在多个样本运行 (Running on multiple samples)
函数FindConservedMarkers()一次只接受一个类群,我们可以在有类群的情况下多次运行这个函数。然而,这样做的效率并不高。相反,我们将首先创建一个函数来查找保守的标记物,包括我们想要包含的所有参数。我们还将添加几行代码来修改输出。我们的函数将:
- 运行
FindConservedMarkers()函数 - 使用
rownames_to_column()函数将行名转移到列中。 - 合并注释
- 使用
cbind()函数创建类群ID列
# 创建用来获取给定类群保守标记物的函数
get_conserved <- function(cluster){
FindConservedMarkers(seurat_integrated,
ident.1 = cluster,
grouping.var = "sample",
only.pos = TRUE) %>%
rownames_to_column(var = "gene") %>%
left_join(y = unique(annotations[, c("gene_name", "description")]),
by = c("gene" = "gene_name")) %>%
cbind(cluster_id = cluster, .)
}现在我们已经创建了这个函数,我们可以把它作为参数用于相应的map函数。我们希望map函数家族的输出是一个数据框,每个类群的输出都由行绑定在一起,我们将使用map_dfr()函数。
map家族的语法:
map_dfr(inputs_to_function, name_of_function)现在,让我们来试试这个函数,找到未识别细胞类型的类群的保守标记物:类群7和类群20。
# 在需要的类群中迭代函数
conserved_markers <- map_dfr(c(7,20), get_conserved)为所有类群寻找标记物对于你的数据,你可能想在所有的类群上运行这个函数,在这种情况下,你可以输入0:20而不是c(7,20);但是,这个函数的运行需要相当长的时间。另外,当你在所有的类群上运行这个函数时,在某些情况下,你可能会有一些类群没有足够多的细胞,而导致函数运行失败。对于这些类群,你需要使用FindAllMarkers()。
评估标记物基因
我们想用这些基因列表来确定这些类群的细胞类型。让我们来看看每个群组排名靠前的基因,看看能不能给我们一些提示。我们可以按两组之间的平均倍数变化来查看每个类群的前10个标记,以便快速浏览。
# 提取每个类群排名靠前的10个标记物
top10 <- conserved_markers %>%
mutate(avg_fc = (ctrl_avg_log2FC + stim_avg_log2FC) /2) %>%
group_by(cluster_id) %>%
top_n(n = 10,
wt = avg_fc)
# 查看每个类群排名靠前的10个标记物
View(top10)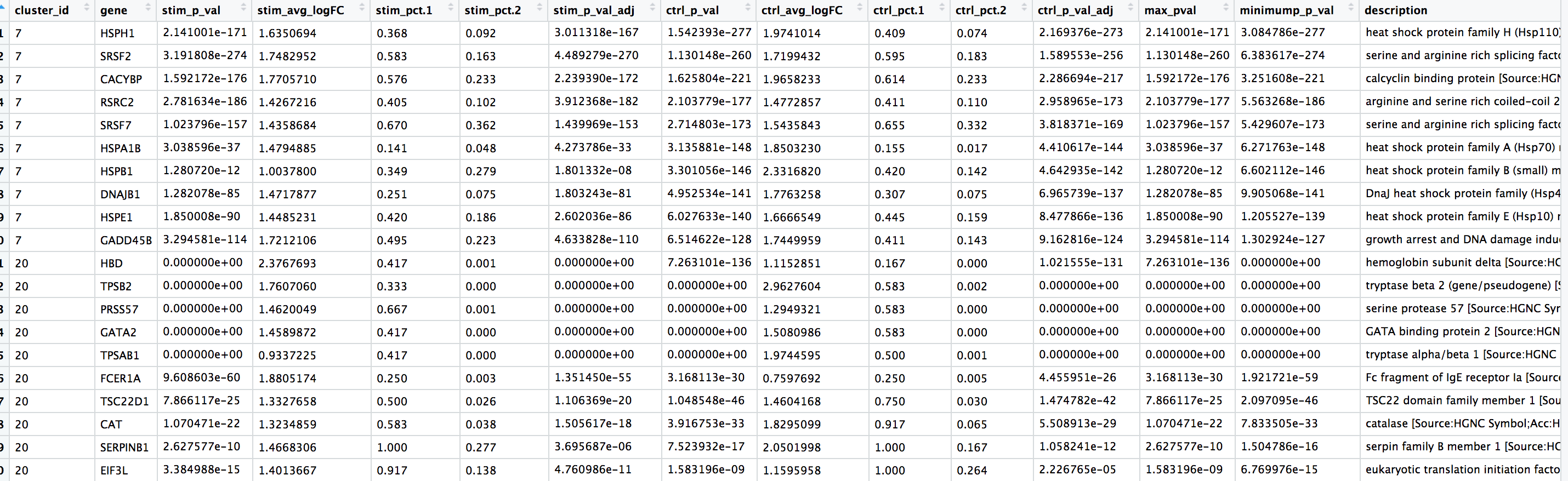
我们看到大量的热休克和DNA损伤基因出现在类群7中。根据这些标记,这些细胞很可能是受压或濒死的细胞。然而,如果我们更详细地探索这些细胞的质量指标(比如该类群的mitoRatio和nUMI),我们并没有看到支持这种说法的有力证据。如果我们更详细地观察基因列表,会发现也有一些T细胞相关的基因和激活的标记物。有可能这些是激活(细胞毒性)T细胞。有广泛的研究支持热休克蛋白与反应性T细胞在慢性炎症中诱导抗炎细胞因子的相关联。为了确定这个类群的身份,我们需要对免疫细胞有更深入的了解,才能真正把结果弄清楚并做出最后的结论。
对于第20号类群,富集的基因似乎并没有一个共同的主题,让我们觉得很突兀。我们通常会看那些pct.1相对于pct.2差异较大的基因,来寻找好的标记基因。比如说,我们可能会对TPSB2这个基因感兴趣,在这个基因群中,有很大一部分细胞表达这个基因,但其他群中表达这个基因的细胞却很少。如果我们 "Google" TPSB2,我们可以在GeneCards网站上找到以下文字:
"β-色氨酸酶似乎是肥大细胞中表达的主要同工酶,而在嗜碱性细胞中,α-色氨酸酶占主导地位。色氨酸酶已被视为哮喘和其他过敏性和炎症性疾病发病机制中的媒介物。"
因此,第20号类群有可能代表肥大细胞 (mast cells)。肥大细胞是免疫系统的重要细胞,属于造血系统。研究发现肥大细胞标志物中明显富含丝氨酸蛋白酶,如TPSAB1和TPSB2,这两个基因都出现在我们的保守标记物列表中。另一个不是丝氨酸蛋白酶,但却是已知的肥大细胞特异性基因,并显示在我们的列表中的基因 ——FCER1A(编码IgE受体的一个亚单位)。此外,我们看到GATA1和GATA2也出现在我们的列表中,它们不是肥大细胞标记基因,但在肥大细胞中大量表达,并且是已知的转录因子,调节各种肥大细胞特异性基因。
观察标记物基因 (Visualizing marker genes)
为了更好地了解第20号聚类的细胞类型识别,我们可以通过使用FeaturePlot()函数探索不同识别的标记物的表达:
# 绘制20号类群的感兴趣标记物基因的表达
FeaturePlot(object = seurat_integrated,
features = c("TPSAB1", "TPSB2", "FCER1A", "GATA1", "GATA2"),
sort.cell = TRUE,
min.cutoff = 'q10',
label = TRUE,
repel = TRUE)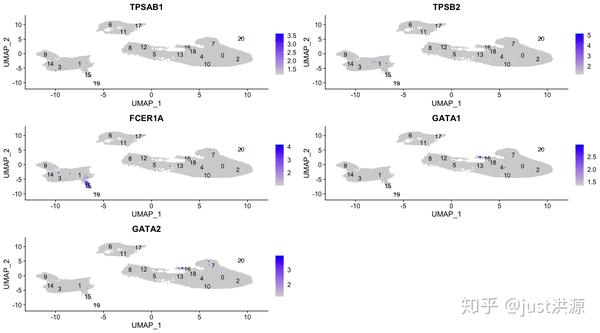
我们还可以通过使用小提琴图 (violin plots)来探索特定标志物的表达范围。
小提琴图类似于方块图,只是它们也显示了不同数值下的数据的概率密度,通常是通过内核密度估计器进行平滑处理。小提琴图比普通的盒形图更有信息量。盒形图只显示总体统计,如均值/中位数和区间范围,而小提琴图则显示数据的完整分布。当数据的分布是多模态的(不止一个峰值)时,这种差异特别有用。在这种情况下,小提琴图显示了不同峰的存在、位置和相对振幅。
# 小提琴图 - 第20号类群
VlnPlot(object = seurat_integrated,
features = c("TPSAB1", "TPSB2", "FCER1A", "GATA1", "GATA2"))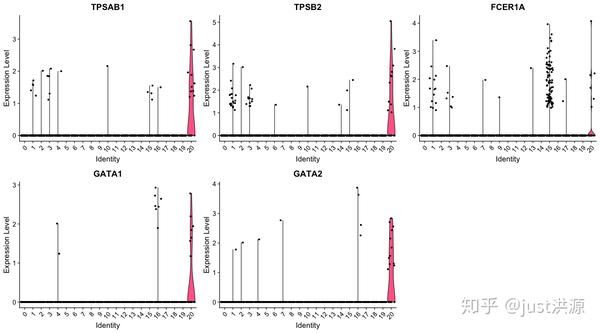
这些结果和图谱可以帮助我们确定这些类群的身份,或者验证在探索了预期的细胞类型的典型标记物后,类群之前假定的身份是否正确。
识别每个类群的基因标记物 (Identifying gene markers for each cluster)
我们最后一组关于分析的问题涉及到相同细胞类型对应的聚类是否具有生物学意义的差异。有时,返回的标记物列表并不能充分分离出一些聚类。例如,我们之前将0、2、4、10和18号聚类群识别为CD4+ Tcells,但这些聚类群的细胞之间是否存在生物学意义上的差异?我们可以使用FindMarkers()函数来确定两个特定的聚类之间有差异表达的基因。
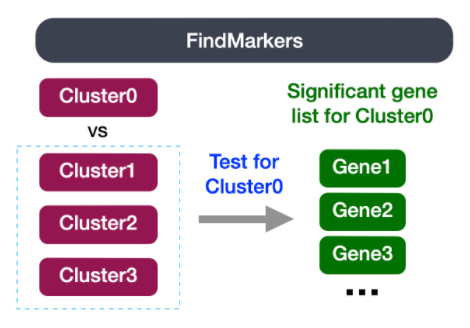
我们可以尝试所有的比较组合,但我们将从类群2与其他所有CD4+ T细胞群的比较开始。
# Determine differentiating markers for CD4+ T cell
cd4_tcells <- FindMarkers(seurat_integrated,
ident.1 = 2,
ident.2 = c(0,4,10,18))
# Add gene symbols to the DE table
cd4_tcells <- cd4_tcells %>%
rownames_to_column(var = "gene") %>%
left_join(y = unique(annotations[, c("gene_name", "description")]),
by = c("gene" = "gene_name"))
# Reorder columns and sort by padj
cd4_tcells <- cd4_tcells[, c(1, 3:5,2,6:7)]
cd4_tcells <- cd4_tcells %>%
dplyr::arrange(p_val_adj)
# View data
View(cd4_tcells)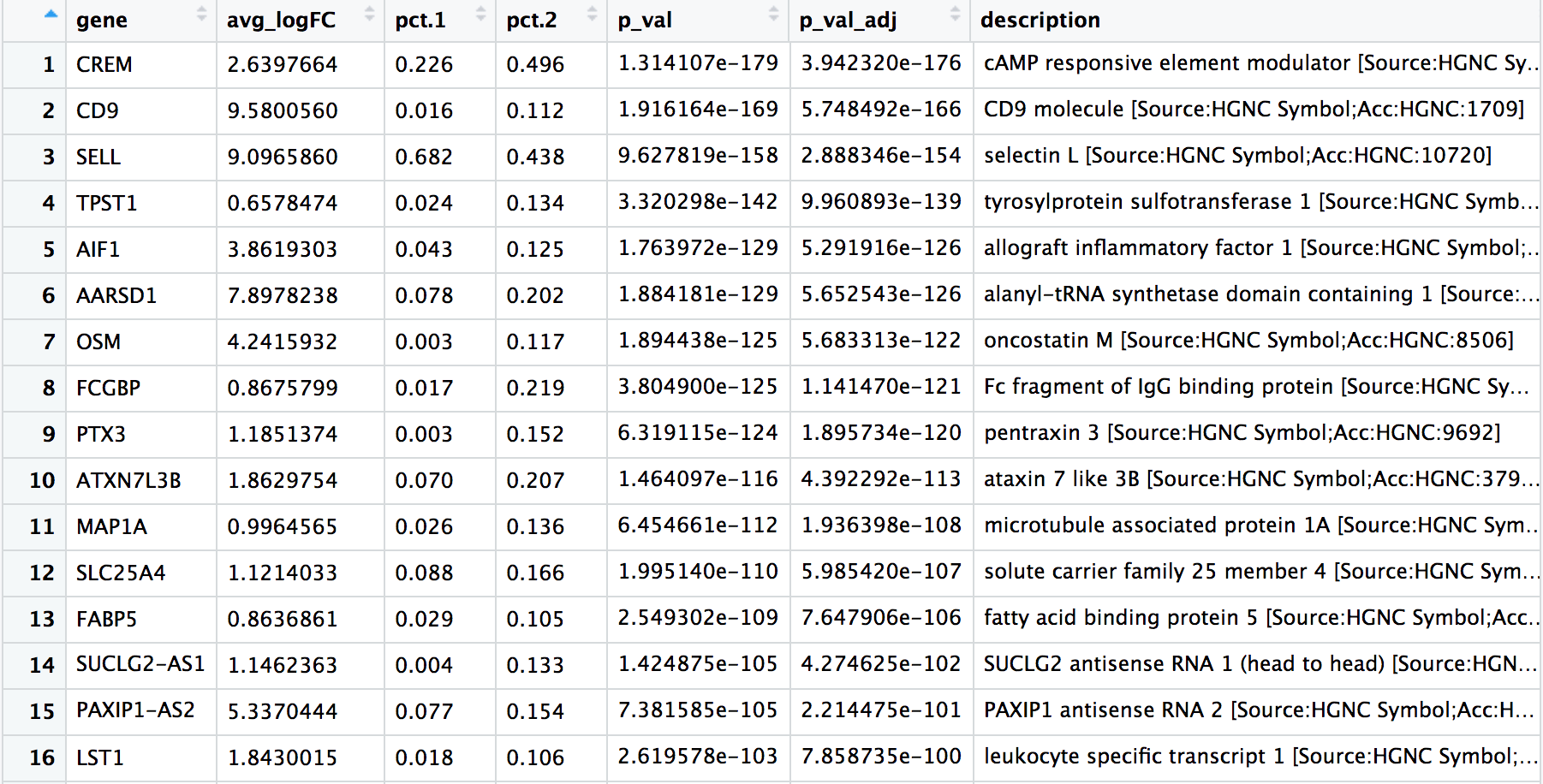
在这些前列基因中,CREM基因作为激活的标志物排在第一位。我们知道,另一个激活的标志物是CD69,初始细胞 (naive cells) 或记忆细胞的标志物包括SELL基因和CCR7基因。有意思的是,SELL基因也排在了榜首。让我们用这些新的细胞状态标记物来直观地探讨一下激活状态。
# Plot gene markers of activated and naive/memory T cells
FeaturePlot(seurat_integrated,
reduction = "umap",
features = c("CREM", "CD69", "CCR7", "SELL"),
label = TRUE,
sort.cell = TRUE,
min.cutoff = 'q10',
repel = TRUE
)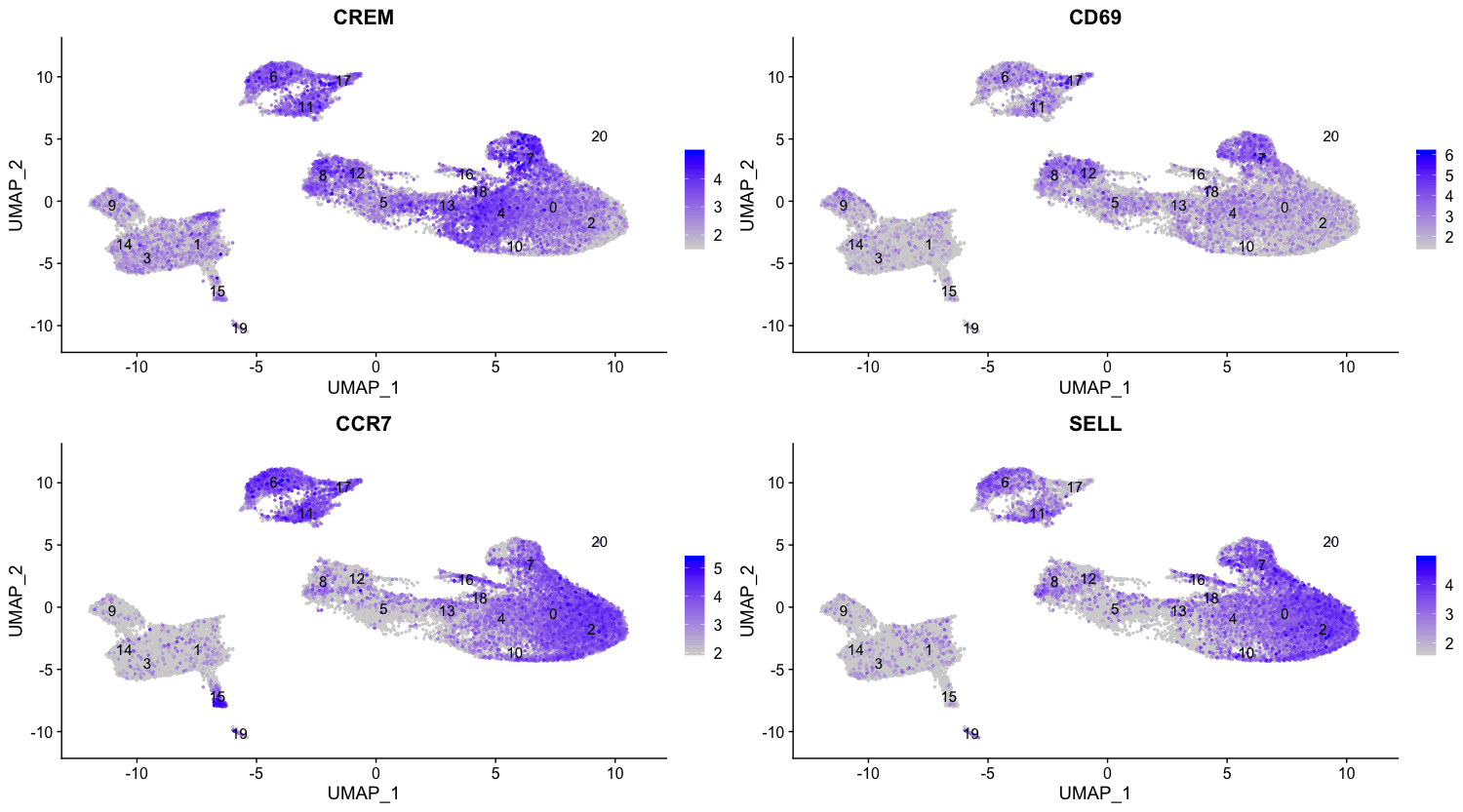
由于标记物列表中同时显示出了原始状态和激活状态的标记物,这对可视化表达很有帮助。根据这些图,似乎0和2号细胞群是可靠的非激活T细胞。然而,对于被激活的T细胞来说,很难说清楚。我们可以说,细胞群4和18是激活的T细胞,但CD69的表达并不像CREM那样明显。我们将标记为初始细胞,剩下的类群标记为CD4+ T细胞。
现在根据所有这些信息,我们可以推测出不同细胞群的细胞类型,然后用细胞类型的标签来绘制细胞。

然后,我们可以将类群的身份重新分配给这些细胞类型:
# 重命名所有类群的身份
seurat_integrated <- RenameIdents(object = seurat_integrated,
"0" = "Naive or memory CD4+ T cells",
"1" = "CD14+ monocytes",
"2" = "Naive or memory CD4+ T cells",
"3" = "CD14+ monocytes",
"4" = "CD4+ T cells",
"5" = "CD8+ T cells",
"6" = "B cells",
"7" = "Stressed cells / Activated T cells",
"8" = "NK cells",
"9" = "FCGR3A+ monocytes",
"10" = "CD4+ T cells",
"11" = "B cells",
"12" = "NK cells",
"13" = "CD8+ T cells",
"14" = "CD14+ monocytes",
"15" = "Conventional dendritic cells",
"16" = "Megakaryocytes",
"17" = "B cells",
"18" = "CD4+ T cells",
"19" = "Plasmacytoid dendritic cells",
"20" = "Mast cells")
# 绘制UMAP图
DimPlot(object = seurat_integrated,
reduction = "umap",
label = TRUE,
label.size = 3,
repel = TRUE)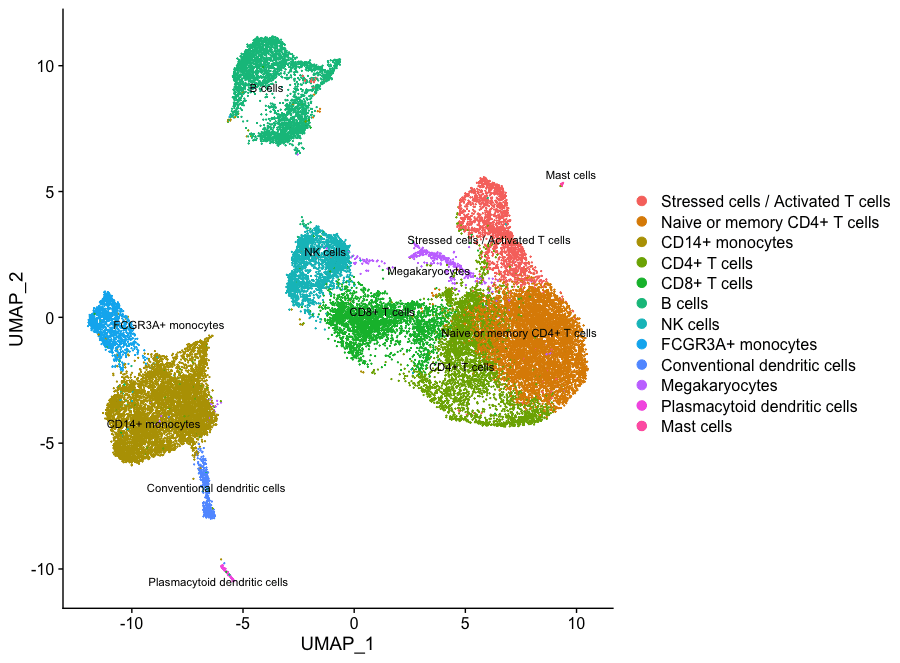
如果我们想删除可能受压的细胞,可以使用subset()函数:
# Remove the stressed or dying cells
seurat_subset_labeled <- subset(seurat_integrated,
idents = "Stressed cells / Activated T cells", invert = TRUE)
# Re-visualize the clusters
DimPlot(object = seurat_subset_labeled,
reduction = "umap",
label = TRUE,
label.size = 3,
repel = TRUE)
现在,我们要保存我们最终标记的Seurat对象:
# Save final R object
write_rds(seurat_integrated,
path = "results/seurat_labelled.rds")
# Create and save a text file with sessionInfo
sink("sessionInfo_scrnaseq_Oct2020.txt")
sessionInfo()
sink()












暂无评论内容