在进行细胞聚类时有两个参数的选择对下游分析的结果影响很大
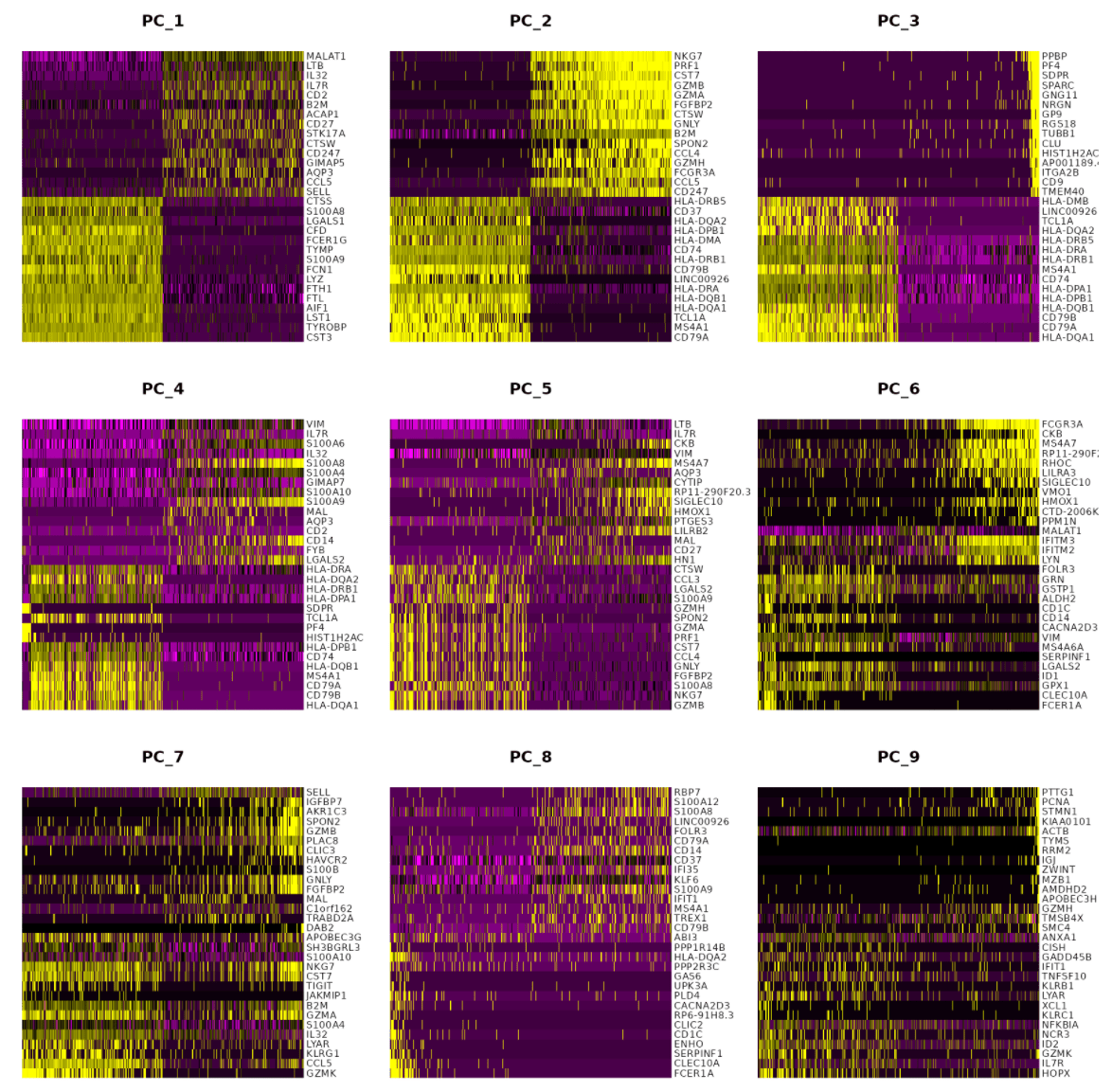
一个是纳入分析的PCs数,也就是dims参数,选择方法见 关于“数据的维度”(dims参数)的选择 ;
另一个是resolution参数,官方推荐根据测到的细胞数进行选择,但是其选择没有具体的标准,可以使用clustree包进行可视化,可以遍历不同resolution取值时细胞分群情况作为参考。
# Check clustering stability at given resolution
# Set different resolutions
res.used <- seq(0.1,1,by=0.2)
res.used
# Loop over and perform clustering of different resolutions
for(i in res.used){
sce <- FindClusters(object = sce, verbose = T, resolution = res.used)
}
# Make plot
library(clustree)
clus.tree.out <- clustree(sce) +
theme(legend.position = "bottom") +
scale_color_brewer(palette = "Set1") +
scale_edge_color_continuous(low = "grey80", high = "red")
clus.tree.out
© 版权声明
文章版权归作者所有,转载请注明来源。
THE END










暂无评论内容