参考链接: https://github.com/hbctraining/scRNA-seq_online/blob/master/lessons/01_intro_to_scRNA-seq.md
我们为什么需要单细胞RNA测序
人类各种组织之间细胞的类型,状态和相互作用差异巨大。而单细胞RNA测序(scRNA-seq)技术提供了在单细胞水平观测基因表达的方法,可以更好地研究这些组织及其中存在的不同类型的细胞。

这一尖端且激动人心的技术可被用于:
- 研究一个组织中到底存在哪些种类的细胞
- 识别未知或少见的细胞类型或状态
- 阐明在分化过程或时间及状态变化中基因表达的改变
- 找出在不同条件下(如加药组和疾病组)在某一特定类型的细胞中差异表达的基因
- 探究一种细胞类型之间基因表达的变化,可同时纳入空间,调控和/或蛋白质信息
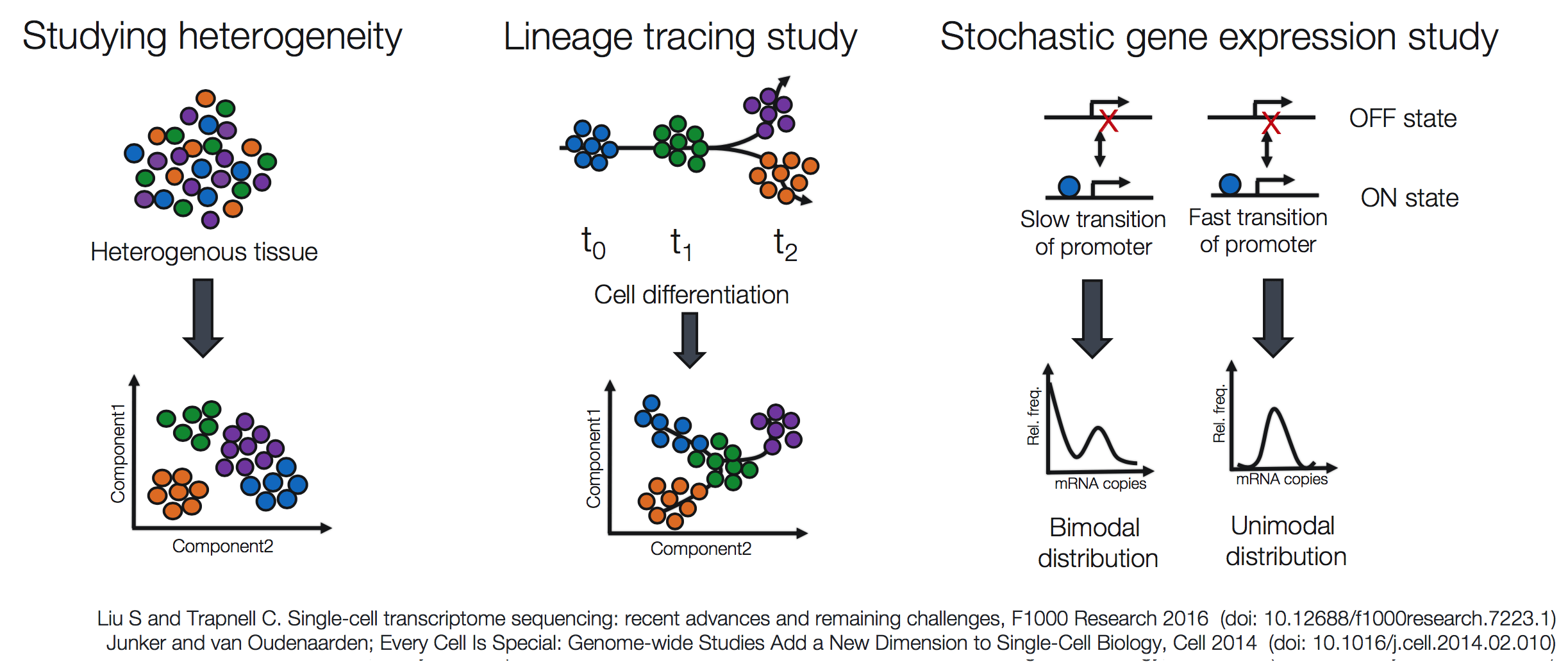
单细胞测序中解决一些较常见问题的方法包括:
- 探究异质性 (Studying heterogeneity)
- 谱系追踪研究 (Lineage tracing study)
- 随机基因表达研究 (Stochastic gene expression study)
单细胞RNA测序分析中面临的挑战
在单细胞RNA测序(scRNA-seq)出现之前,转录组分析是使用批量RNA测序(bulk RNA-seq)进行的,这是一种比较细胞基因表达平均值 (averages of cellular expression)的直接方法。如果要寻找疾病的生物学标志物 (disease biomarkers),或者不关心样本中的细胞异质性,这可能也是很好的方法。
虽然批量RNA测序可以探索不同条件(如加药或疾病)之间基因表达的差异,但在细胞水平上的差异并没有被充分挖掘。例如,在下图的展示中,如果进行批量(bulk)分析(左),我们将无法检测到基因A和基因B表达之间的正确关系。但是,如果我们按照细胞类型或细胞状态对细胞进行适当分群,我们可以看到基因之间的正确关系。
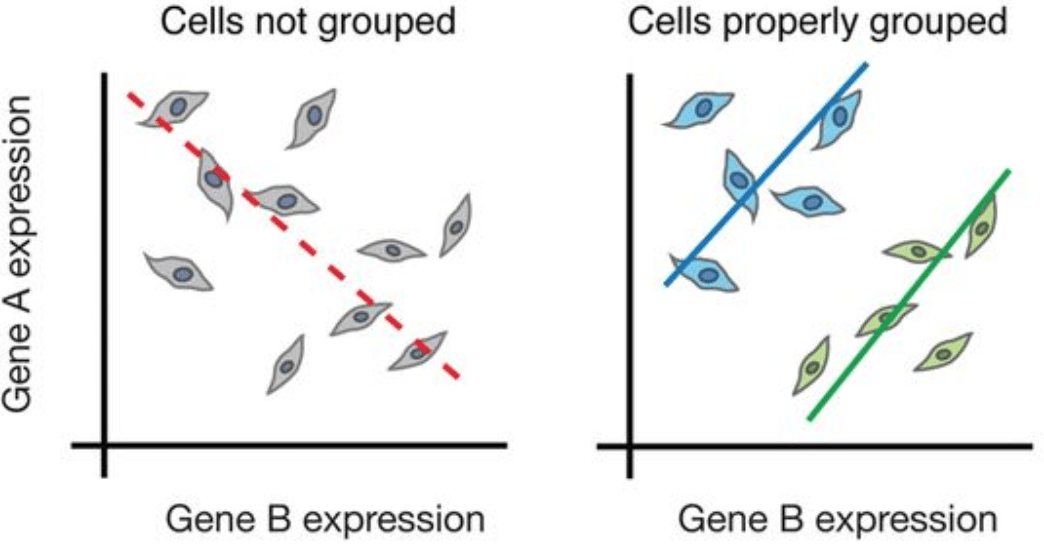
图片来源:Trapnell, C. Defining cell types and states with single-cell genomics, Genome Research 2015
尽管单细胞RNA测序能够获得在细胞水平上的基因表达,但样本制备和建库成本更高,分析也更复杂,更难解读。单细胞RNA测序分析的复杂性包括:
- 庞大的数据量
- 每个细胞测序深度低
- 跨细胞/样本的生物学误差
- 跨细胞/样本的技术性误差
庞大的数据量
单细胞RNA测序项目的表达量数据代表了成千上万个细胞中上万或数十万条读取 (reads),产生的数据要大得多,需要更多的内存来分析,更大的硬盘来存储,以及更多的时间来运行分析。
每个细胞测序深度低
对于基于油滴(droplet)的单细胞RNA测序方法,测序的深度较浅,通常每个细胞只能检测到10-50%的转录组。这导致细胞中许多基因显示为零计数。然而,在单个细胞中,一个基因的零计数未必表示该基因没有被表达,也有可能只是因为表达量低而没有检测到转录本。从总体来看,表达水平较高的基因零计数的细胞就更少。由于这一特性,许多基因在任何细胞中都不会被检测到,细胞间的基因表达也会有很大的差异。
跨细胞/样本的生物学误差
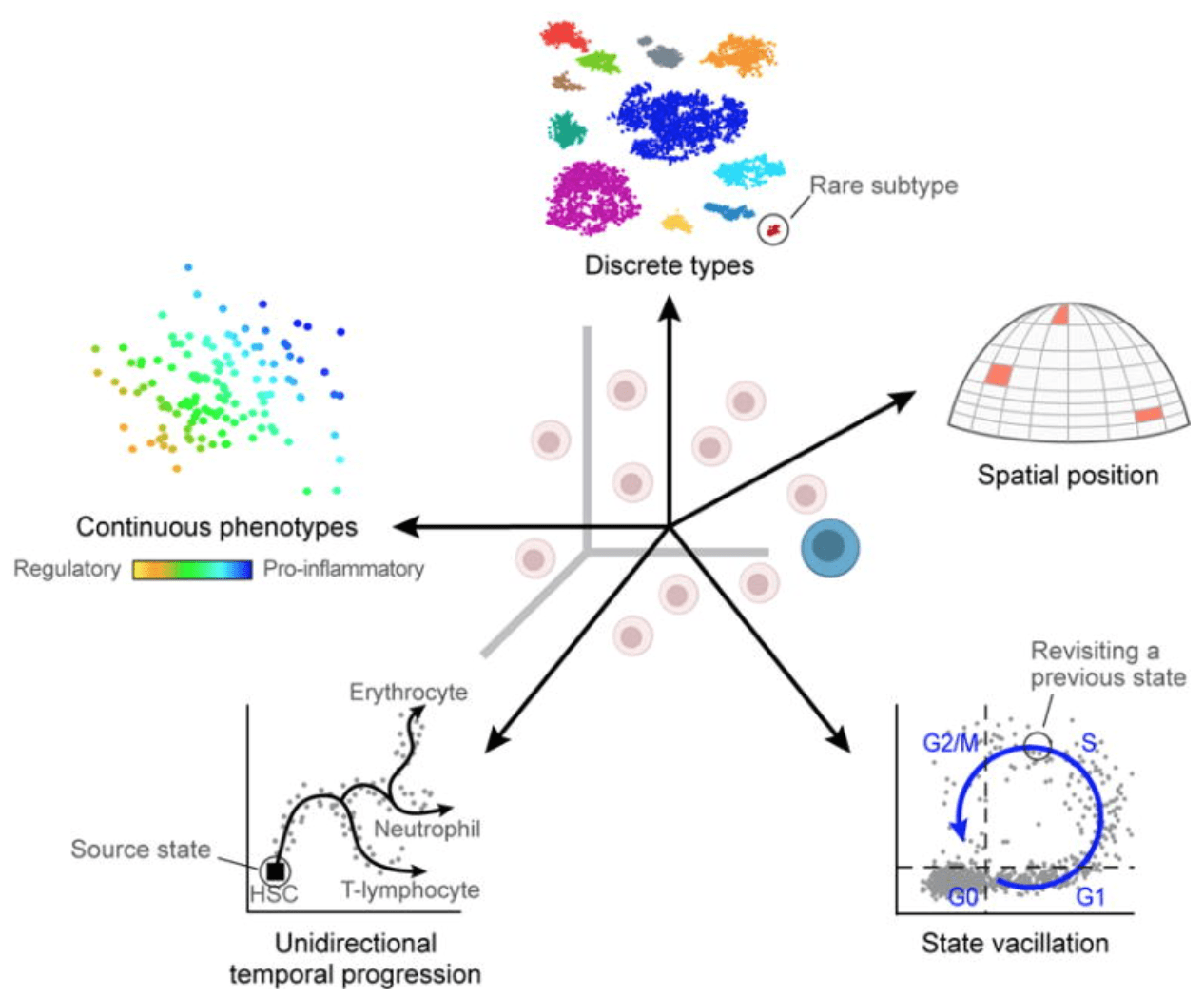
我们不感兴趣的生物学变异会导致细胞间的基因表达跟实际的生物细胞类型或状态更为相似或不同,从而影响细胞类型的识别。这些生物学变异(除非是实验研究的一部分)包括:
- 转录爆发? (Transcriptional Bursting):并非所有基因的转录过程始终处于开启状态。收样的时间点将决定每个细胞中的基因是否处在转录的状态。(理解为基因转录的过程不是连续不断的,而是脉冲式的,尽管某个条件下某种细胞会表达某些基因,但是收样的时候这些细胞有可能处于转录的间歇期)
- RNA加工的不同速度 (Varying rates of RNA processing):不同的RNA以不同的速率进行加工。
- 连续或离散的细胞特征 (Continuous or discrete cell identities):例如单个T细胞的促炎症潜能。我们使用基因的差异表达来定义不同的细胞状态,但是实际上细胞的状态变化是一个连续的过程,使用离散特征描述连续过程存在困难。
- 环境刺激 (Environmental stimuli):细胞的周边环境可以通过空间位置、信号分子等方式影响基因的表达。
跨细胞/样本的技术性误差
除了细胞类型或状态,技术来源的变异也会使基因表达谱产生差异,这会影响细胞类型的识别。技术来源的变异包括:
- 不同细胞的捕获效率 (Cell-specific capture efficiency):不同细胞捕获的转录本数量不同,导致测序深度不同(例如,转录组的10-50%)。
- 文库质量 (Library quality):降解的RNA、低活性/濒死的细胞、大量游离的RNA、分离不好的细胞以及细胞定量不准确都会导致测序质量过低。
- 扩增偏差 (Amplification bias):在建库(library preparation)的扩增步骤中,并非所有的转录本都被扩增到相同的水平。
- 批次差异 (Batch effects):批次问题是单细胞RNA分析中的一个重要问题,下图可以看到由于批次问题而导致的基因表达上的显著差异。

图片来源:Hicks SC, et al., bioRxiv (2015) 这些研究设计中由于批次处理不当所产生的问题在这篇文章中有着精彩的阐述。
如何知道你的实验是否存在批次问题?
- 所有的RNA提取都是在同一天进行的吗?
- 所有的建库工作都是在同一天进行的吗?
- 所有样品的RNA提取或建库是否由同一个人完成?
- 你是否对所有样品都使用了相同的试剂?
- 你是否在同一地点进行了RNA的提取或建库?
如果这些答案中有一个是“不”,那么就有批次问题。
有关批次问题的最佳实践
- 在实验设计中尽可能地避免产生批次
- 如果无法避免批次:
- 不要混淆不同批次的实验
- 请在不同批次中做不同类别样品的重复(replicates)。如果是想找出不同处理条件下的差异基因或者在群体水平上下结论,重复自然越多越好(当然要大于2)。如果使用一次实验只建库一次的
inDrops方法,则应各组样本交替建库(例如,不要先建所有对照组的文库,然后再建所有处理组的文库)。 - 请在实验的元数据(metadata)中包含批次信息。这样在分析中我们就可以去除由于批次或者多批次聚合(integrate)产生的差异。当我们有这些信息的话就不会影响我们最终的结果。
总结
虽然单细胞RNA测序是一种强大而深刻的从单细胞水平分析基因表达的方法,但由于存在许多挑战和变异因素,单细胞转录组的数据分析显得复杂且受限。
综上所述,我们提出以下建议:
- 除非对解决我们感兴趣的科学问题有必要,否则不要进行单细胞RNA测序。你是否能够使用更简单,更廉价的批量RNA测序解决你的科学问题?也许可以使用流式细胞仪(FACS)对样本进行分选,以便进行批量分析?
- 了解你想解决的科学问题的各种细节。推荐的建库方法和分析流程可以根据具体实验而有所不同。
- 尽可能地避免技术来源的差异:
- 实验开始前与专家讨论实验设计
- 同时从样品中提取RNA
- 同时建库或替换样本组以避免批次混淆
- 不要混淆不同性别、年龄和批次的样本组










暂无评论内容