在我们我们获得高质量的单细胞数据后,单细胞RNA测序分析工作流程的下一步是进行聚类。聚类的目标是将不同的细胞类型分离成独特的细胞群。为了执行聚类,我们需要确定细胞间表达差异最大的基因。然后,我们使用这些基因来确定哪些相关的基因集在细胞之间的表达差异最大。
然而,在我们进入聚类之前,有几个概念需要了解一下。
计数归一化 (Count normalization)
首先是计数归一化,这对于准确比较细胞(或样品)之间的基因表达至关重要。每个基因的映射读取的计数值与RNA的表达量("有意义的")以及许多其他因素("无意义的")成正比。归一化是考虑到 "无意义"的因素而对原始计数值进行缩放的过程。通过这种方式,细胞之间和/或细胞内的表达水平更具有可比性。
归一化过程中经常考虑的主要因素有。
- 测序深度 (Sequencing depth):考虑测序深度对于细胞间基因表达的比较是必要的。在下面的例子中,每个基因在细胞2中的表达量似乎都增加了一倍,然而这是细胞2具有两倍的测序深度的结果。单细胞RNA测序中的每个细胞都会有不同数量的相关读取。因此,为了准确比较细胞之间的表达,需要对测序深度进行归一化。
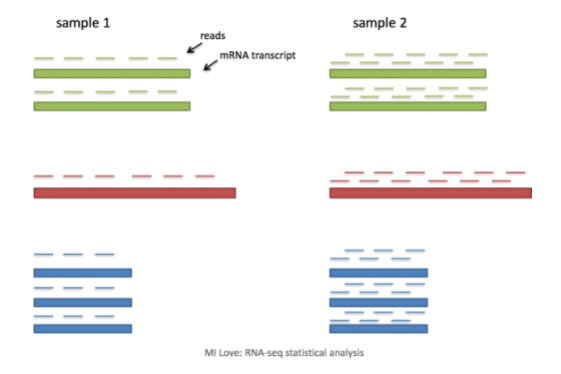
- 基因长度 (Gene length):基因长度的考量对于比较同一细胞内不同基因之间的表达是必要的。映射到一个较长的基因上的读取可能与较短的表达量较高的基因一样,看起来有相等的读取/表达量。在单细胞RNA测序分析中,我们将比较细胞内不同基因的表达情况,对细胞进行聚类。如果使用基于3’或5’端的液滴法,基因的长度不会影响分析,因为只对转录本的5’或3’端进行测序。但是,如果使用全长测序法,应考虑到转录本的长度。
主成分分析 (PCA)
主成分分析(PCA)是一种用于强调变异性和相似性,并在数据集中找出突出特征的技术(降低维度)。我们将在本文中简要介绍一下PCA(改编自StatQuests/Josh Starmer的YouTube视频),但强烈建议你去研究StatQuest的视频,以获得更全面的解释/理解。
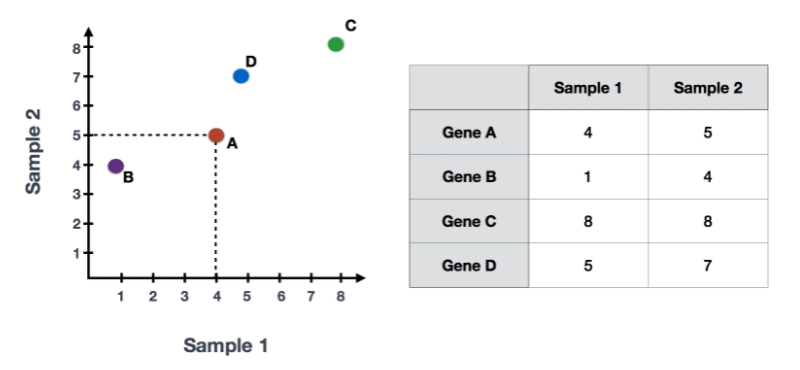
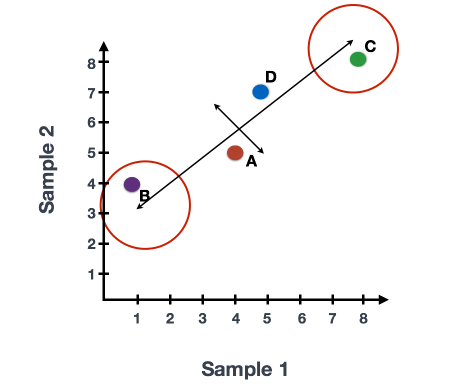
如果你在两个样本(或细胞)中量化了四个基因的表达,你可以用一个样本在x轴上表示,另一个样本在y轴上表示这些基因的表达值,如下图所示。
你可以在代表变异最大的方向上画一条线贯穿数据,在这个例子中是在对角线上。数据集中的最大变异是在这条线的两个端点的基因之间。
我们还可以看到基因在这条线的上方和下方有一定程度的变化。我们可以通过数据画另一条线,代表数据中的第二大变异量,因为这个图是2D(2轴)。
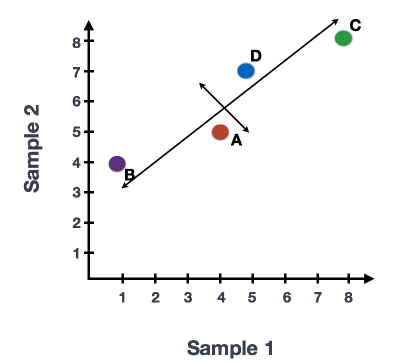
每条线的两端附近的基因将是那些变异最大的基因;这些基因在数学上对直线的方向影响最大。
例如,基因C的值稍有变化,就会极大地改变长线的方向,而基因A或基因D改变很小,对其影响不大。
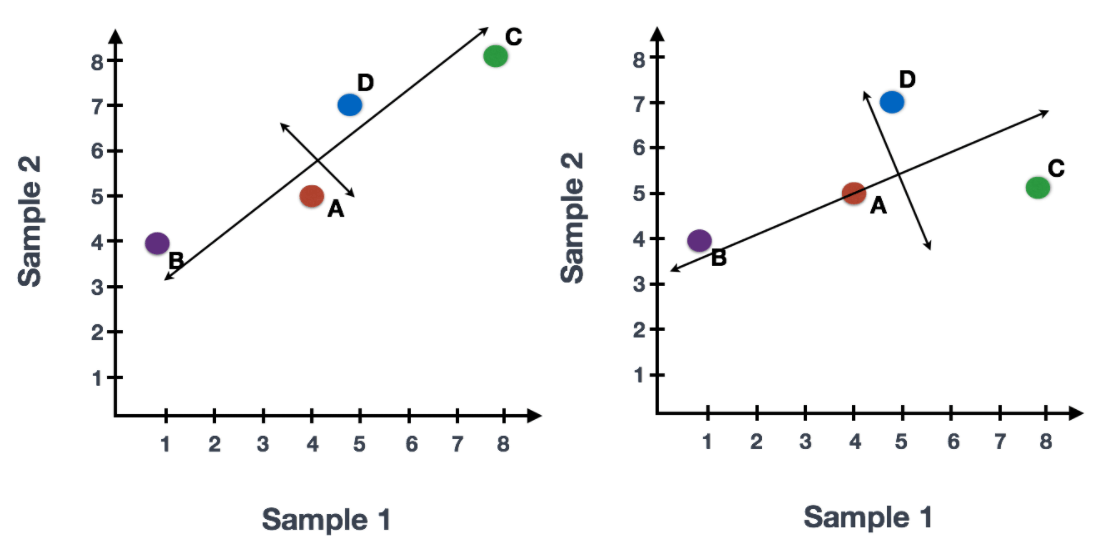
我们还可以旋转整个图像,将代表变化的线条看成从左至右和从上至下的变化。我们可以看到数据中的大部分变化是由左至右(长线),其次是上下变化(短线)。现在你可以把这些线看作是代表变化的轴。这些轴基本上就是 "主成分",PC1代表数据中变化最大的数据,PC2代表数据中变化第二大的数据。
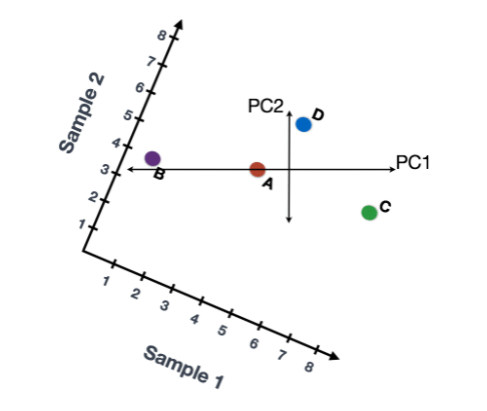
如果我们有三个样本/细胞,那么我们将有一个额外的方向,在这个方向上我们可以有变化(3D)。因此,如果我们有N个样本/细胞,我们将有N个变异方向或主成分(PC)! 一旦这些PC被计算出来,处理数据集中变异最大的PC被指定为PC1,下一个被指定为PC2,以此类推。
一旦确定了一个数据集的PC,我们就必须弄清楚每个样本/细胞如何与这个背景相吻合,以使得我们能够以直观的方式显示出相似性/差异性。这里的问题是 "基于样本X中的基因表达,样本X对于给定PC的得分是多少?"。按照以下步骤计算所有样本-PC对的得分。
- 首先,根据每个基因对每个PC的影响程度,给每个基因分配一个 "影响 "分数。对给定的PC没有任何影响的基因得到接近于零的分数,而影响较大的基因得到较大的分数。PC线两端的基因会有较大的影响,所以它们会得到较大的分数,但符号相反。
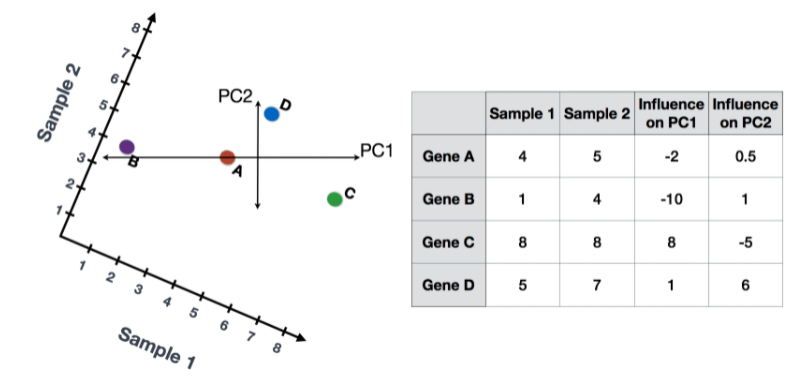
- 确定了影响后,用以下公式计算每个样品的得分:
Sample1 PC1 score = (read count * influence) + ... for all genes对于我们的两样本示例来说,下面将会展示这些分数将如何被计算
## Sample1
PC1 score = (4 * -2) + (1 * -10) + (8 * 8) + (5 * 1) = 51
PC2 score = (4 * 0.5) + (1 * 1) + (8 * -5) + (5 * 6) = -7
## Sample2
PC1 score = (5 * -2) + (4 * -10) + (8 * 8) + (7 * 1) = 21
PC2 score = (5 * 0.5) + (4 * 1) + (8 * -5) + (7 * 6) = 8.5- 一旦这些所有PC的分数被计算出来,就可以在一个简单的散点图上绘制出来。
对于样本或细胞数量较多的数据集,通常会绘制出每个样本/细胞的PC1和PC2分数。由于这些PC解释了数据集中最大的变化,所以我们预期的是,彼此比较相似的样本/细胞将在PC1和PC2上聚集在一起。见下面的例子。
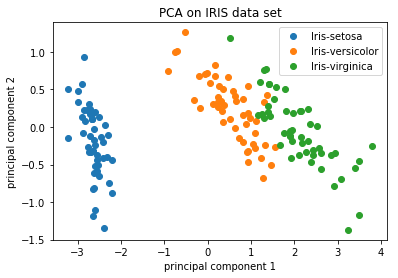
数据来源: https://github.com/AshwiniRS/Medium_Notebooks/blob/master/PCA/PCA_Iris_DataSet.ipynb
有时也会使用较高的PC(如PC3,PC4等)来进行可视化,特别是当PC1和PC2解释的变异不是很大,并且数据集有多个因素导致样本之间的变异时。
对于我们的单细胞RNA测序数据,我们将使用10-100个PC分值来比较所有细胞之间的表达,而不是比较所有细胞中20000多个基因的表达,这将突出我们数据集中最大的变异来源。除了探索与这些最高PC值相关的变异来源,我们还将根据表达的相似性使用这些PC值来聚类我们的细胞。










暂无评论内容