学习内容
- 学会选择合适的PCs用于聚类分析
- 聚类的方法
目标
- 产生细胞类型特异性聚类,并使用已知的细胞类型标记基因来确定聚类的身份。
- 确定集群是否代表真正的细胞类型或由于生物或技术差异而产生的集群,如细胞周期S期的细胞集群、特定批次的集群或线粒体含量高的细胞。
挑战
- 识别出可能是由于生物学或技术上的无意义变化而导致的质量较差的簇
- 识别每个集群的细胞类型
- 保持耐心,因为这可能是一个在聚类和标记物识别之间高度反复的过程(有时甚至要回到QC过滤中去)。
基于首要主成分进行集群
设置
在开始本课程之前,让我们为工作流中的后续几个步骤创建一个名为clustering.R的新脚本。
# Single-cell RNA-seq - clustering
# Load libraries
library(Seurat)
library(tidyverse)
library(RCurl)
library(cowplot)识别有意义的主成分
参考 关于“数据的维度”(dims参数)的选择、 Seurat基本分析流程
为了克服scRNA-seq数据中任何单一基因表达的广泛技术噪音,Seurat根据它们的PCA分数将细胞分配到集群中,PCA分数来自于整合的最易变的基因的表达,每个PC基本上代表一个 "元基因",结合了整个相关基因组的信息。因此,确定在聚类步骤中包括多少个PC是很重要的,以确保我们捕捉到数据集中存在的大部分变异或细胞类型。
在决定要包括哪些PC以便进行下游聚类分析之前,先探索PC是很有用的。
热图
探索PC的一种方法是使用热图来可视化选择PC的最变异的基因,并按PCA分数排序的基因和细胞。这里的想法是看一下PC,并确定驱动它们的基因对区分不同的细胞类型是否有意义。
cells参数指定PCA分数为负数或正数最多的单元数,以用于绘图。我们要找到从某一个PC开始其热图开始看起来更加“模糊”,即基因组之间的区别不太明显。
# Explore heatmap of PCs
DimHeatmap(seurat_integrated,
dims = 1:9,
cells = 500,
balanced = TRUE)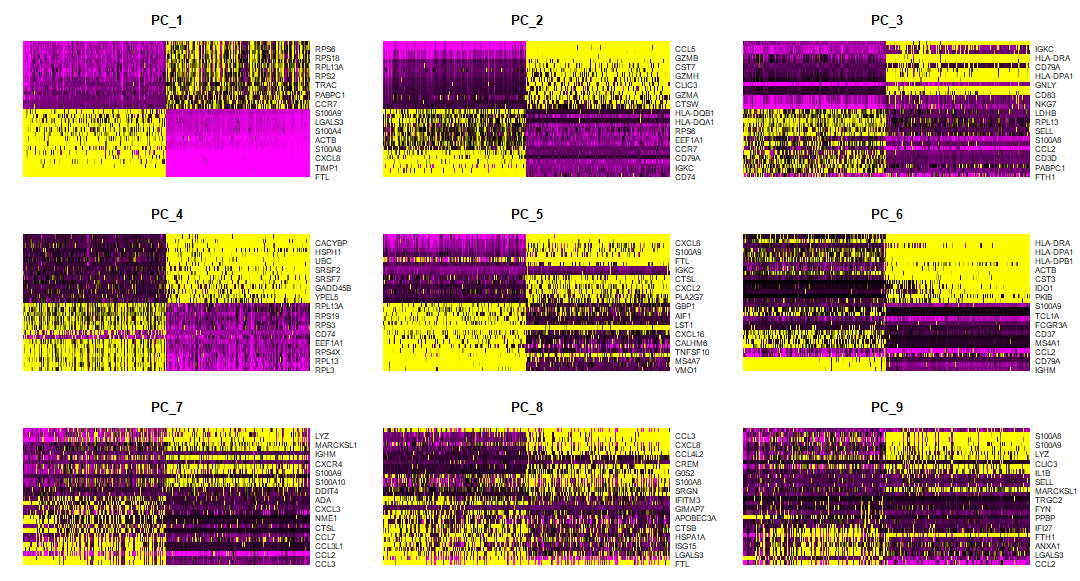
如果我们想探索大量的PC,这种方法可能会很慢,而且很难将单个基因可视化。同样,为了探索大量的PC,我们可以通过驱动PC的PCA得分,打印出前10个(或更多)正负基因。
# Printing out the most variable genes driving PCs
print(x = seurat_integrated[["pca"]],
dims = 1:10,
nfeatures = 5)
碎石图(肘形图?)
碎石图是另一种有帮助的方法,可以确定使用多少个PC进行聚类,以使我们能够捕捉到数据中的大部分变化。肘形图直观地显示了每个PC的标准差,我们正在寻找标准差开始趋于平稳的地方。从本质上讲,肘部出现的地方通常是识别大部分变异的阈值。然而,这种方法可能是相当主观的。
使用前40个PCs绘制碎石图:
# Plot the elbow plot
ElbowPlot(object = seurat_integrated,
ndims = 40)
根据这个图,我们可以通过PC8-PC10左右的形成转角的地方大致确定大部分的变化,或者也有人会认为应该是数据点开始接近X轴的时候,PC30左右。这给了我们一个非常粗略的想法,即需要包括的PC数量,我们可以以更多的量化方式提取这里的可视化信息,这可能更可靠一点。
尽管以上两种方法与来自Seurat的较旧方法一起用于标准化和鉴定可变基因的方法更多,但它们不再像以前那样重要。这是因为SCTransform方法比旧方法更准确。
为什么选择PC对于较旧的方法更重要?
较早的方法将一些技术性的变异来源纳入了一些较高的PC中,因此PC的选择更为重要。SCTransform可以更好地估计方差,并且不经常将这些技术性变异源纳入较高的PC中。
从理论上讲,使用SCTransform,我们选择的PC越多,在进行聚类时就会考虑到更多的变化,但是进行聚类需要花费很多时间。因此,在这次分析中,我们将使用前40个PC来生成聚类。
聚类
Seurat使用基于图形的聚类方法,将细胞嵌入一个图形结构中,使用K-近邻(KNN)图(默认情况下),在具有类似基因表达模式的细胞之间绘制边。然后,它试图将这个图划分为高度相互关联的 "准集群 "或 "社区"。
我们将使用FindClusters()函数来进行基于图形的聚类。分辨率是一个重要的参数,它设定了下游聚类的 "粒度",需要为每个实验进行优化。对于3,000-5,000个细胞的数据集,分辨率设置在0.4-1.4之间通常会产生良好的聚类。增加分辨率值会导致更多的聚类,这对于更大的数据集来说往往是必需的。
FindClusters()函数允许我们输入一系列的分辨率,并将计算出聚类的 "粒度"。这对于测试哪种分辨率适合前进非常有帮助,而不必为每个分辨率运行该函数。
# Determine the K-nearest neighbor graph
seurat_integrated <- FindNeighbors(object = seurat_integrated,
dims = 1:40)
# Determine the clusters for various resolutions
seurat_integrated <- FindClusters(object = seurat_integrated,
resolution = c(0.4, 0.6, 0.8, 1.0, 1.4))如果我们看一下我们的Seurat对象的元数据(seurat_integrated@metadata),有一个单独的列用于计算不同的分辨率。
# Explore resolutions
seurat_integrated@meta.data %>%
View()为了选择一个开始的分辨率,我们通常会选择一些中间的范围,如0.6或0.8。我们将从0.8的分辨率开始,通过使用Idents()函数分配群集的身份。
# Assign identity of clusters
Idents(object = seurat_integrated) <- "integrated_snn_res.0.8"为了可视化细胞簇,有几种不同的降维技术可能会有所帮助。最受欢迎的方法包括t分布随机邻居嵌入(t-SNE)和均匀流形逼近与投影(UMAP)技术。
这两种方法都是为了将高维空间中具有相似局部邻域的细胞放在低维空间中。这些方法需要你输入用于可视化的PCA维数,我们建议使用相同数量的PC作为聚类分析的输入。在这里,我们将继续使用UMAP方法来实现聚类的可视化。
## Calculation of UMAP
## DO NOT RUN (calculated in the last lesson)
# seurat_integrated <- RunUMAP(seurat_integrated,
# reduction = "pca",
# dims = 1:40)# Plot the UMAP
DimPlot(seurat_integrated,
reduction = "umap",
label = TRUE,
label.size = 6)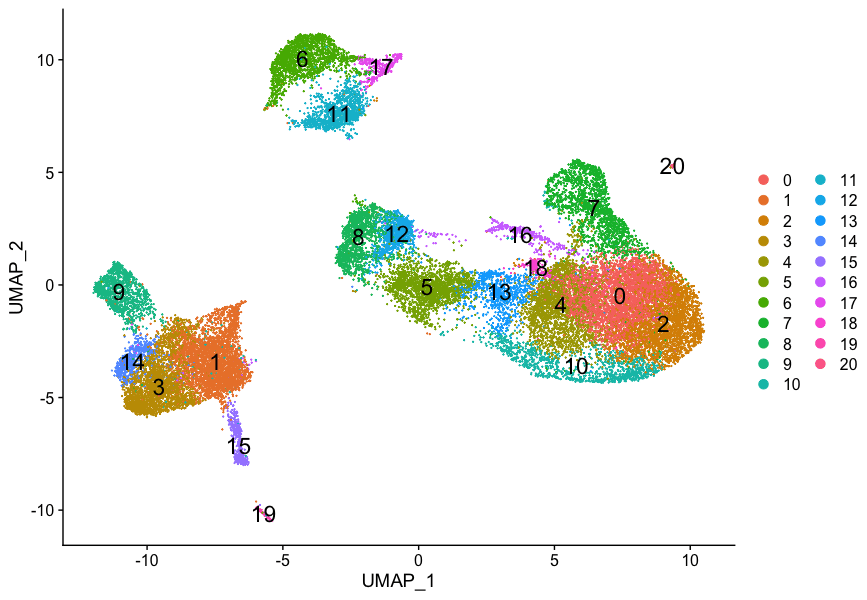
它对于探索其他分辨率也是很有用的。它可以让你快速了解到集群将如何根据分辨率参数而变化。例如,让我们切换到一个0.4的分辨率。
# Assign identity of clusters
Idents(object = seurat_integrated) <- "integrated_snn_res.0.4"
# Plot the UMAP
DimPlot(seurat_integrated,
reduction = "umap",
label = TRUE,
label.size = 6)
实际运行出来的图形分布及聚类可能与教程中的图不太一样,这是因为seurat及相应包的版本不同导致的细微差异。
现在,我们将继续使用0.8分辨率来检查质量控制指标和预期细胞类型的已知标记。再次绘制UMAP,以确保您现在(或仍然)的图像与您在课程中看到的图像匹配:
# Assign identity of clusters
Idents(object = seurat_integrated) <- "integrated_snn_res.0.8"
# Plot the UMAP
DimPlot(seurat_integrated,
reduction = "umap",
label = TRUE,
label.size = 6)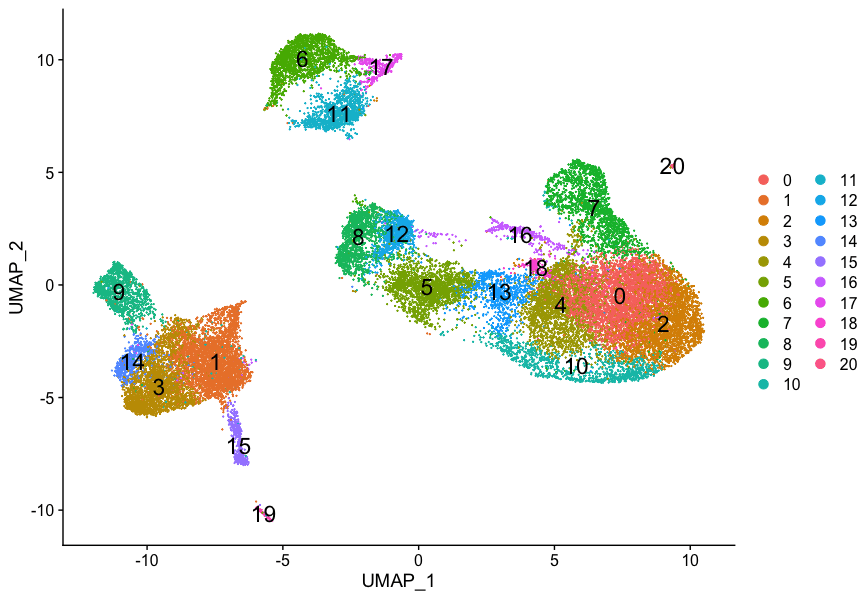











暂无评论内容