1. 下载sra文件(多线程高速版) download_sra.sh
脚本说明:
-
使用前请确认已经正确安装
sra-tools; -
这个脚本是使用
prefetch命令从ncbi的数据库中多线程下载sra文件,不论是bulk-seq还是single cell的原始数据,只要是sra均可使用; -
如果要从EBI下载
fastq,不适用此脚本,建议使用ascp; -
修改变量:
outdir请改成自己的输出目录,sra_id_file改成SRR_Acc_List.txt的路径,max_jobs根据自己情况修改同时下载多少个; -
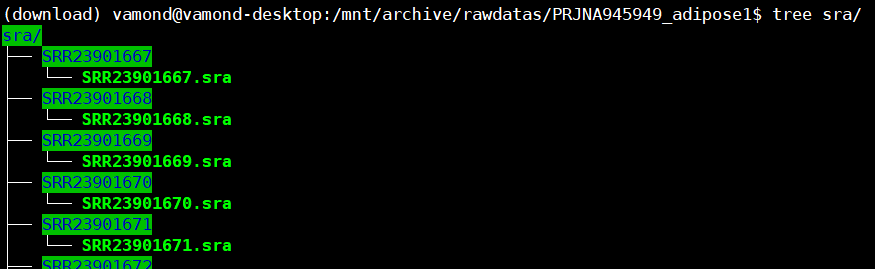
最后输出的文件格式为 $outdir/SRR../SRR…sra

-
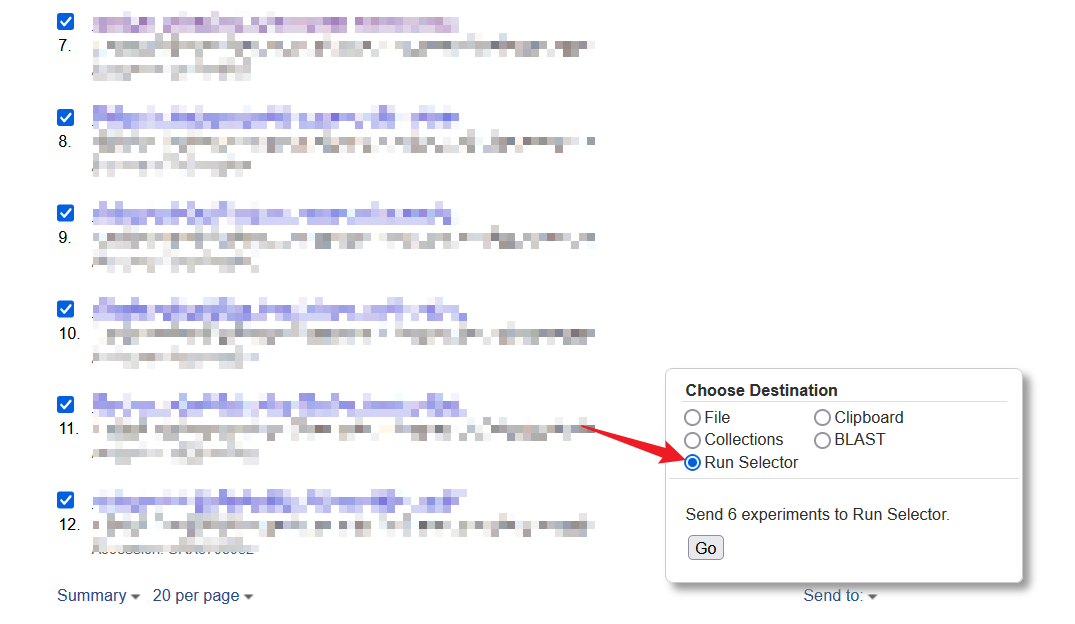
SRR_Acc_List.txt 放在运行脚本的目录即可。该文件是在SRA RUN SELECTOR下载的srr列表,获取方法是找到你想要下载的项目的sra列表,然后页面上有个send to 按钮,选择file-Accession List,生成即可。如果不需要全部的run,可以自己按需删掉部分run:

-
偶尔遇到单个文件没有下载成功可以使用以下命令进行下载:
prefetch -p -X 209715200 -O $outdir $sra_id # 比如: prefetch -p -X 209715200 -O /mnt/archive/rawdatas/PRJNA562645_hs_av/sra SRR25112332
脚本内容及运行命令
# 创建文件
vim download_sra.sh
复制以下脚本粘贴进去,:wq保存
#!/bin/bash
outdir=/mnt/archive/rawdatas/PRJNA___/sra
sra_id_file=SRR_Acc_List.txt
max_jobs=6
if [ ! -d "$outdir" ]; then
mkdir -p $outdir
fi
while read sra_id; do
prefetch -X 209715200 -O $outdir $sra_id &
if (( $(jobs -r -p | wc -l) >= max_jobs )); then
wait -n
fi
done < $sra_id_file
wait
# 我已经提前将下载数据所用的常用软件安装到 conda 环境中了
conda activate download
# 后台运行脚本并输出日志
nohup bash download_sra.sh > download_sra.log 2>&1 &
# 查看日志
tail -f download_sra.log
这篇文章介绍了一个多线程下载NCBI数据库中SRA文件的脚本download_sra.sh。使用前需要确认已安装sra-tools。该脚本通过prefetch命令,可以高效地下载各种SRA文件,包括bulk-seq和single cell原始数据。用户需根据个人需求设置输出目录、SRR ID文件路径及并行下载的最大任务数。下载列表文件(SRR_Acc_List.txt)需在运行脚本同目录下,文件生成方法是通过SRA RUN SELECTOR界面进行相应操作。若单个文件下载失败,可手动使用prefetch命令重试。文中还提供了脚本内容及运行命令,强调在conda环境中执行,并可以后台运作以查看日志。











暂无评论内容