scRNA-seq数据整合Introduction
Seurat不仅可以校正实验的批次效应,还能跨平台整合数据,例如将10x单细胞数据、BD单细胞数据和SMART单细胞数据整合在一起;也能整合单细胞多组学数据,例如将单细胞ATAC、空间转录组与单细胞转...
整合-HBC lesson 6.2
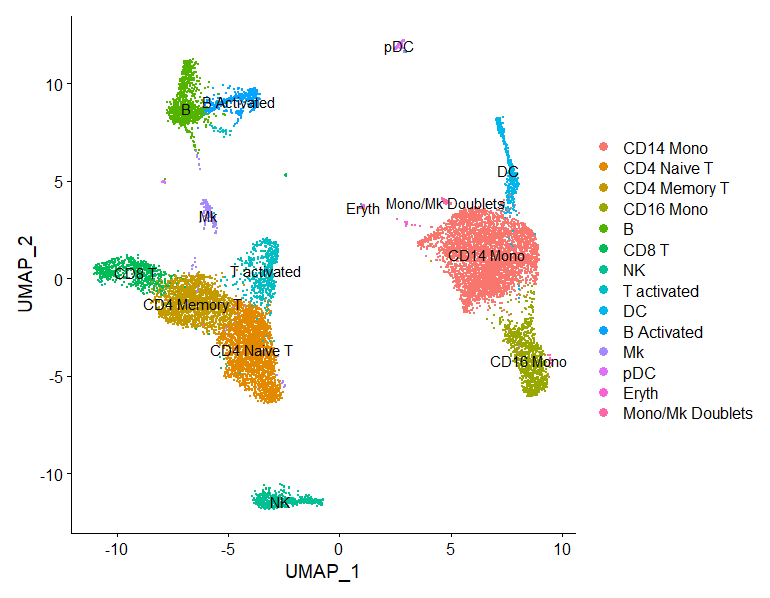
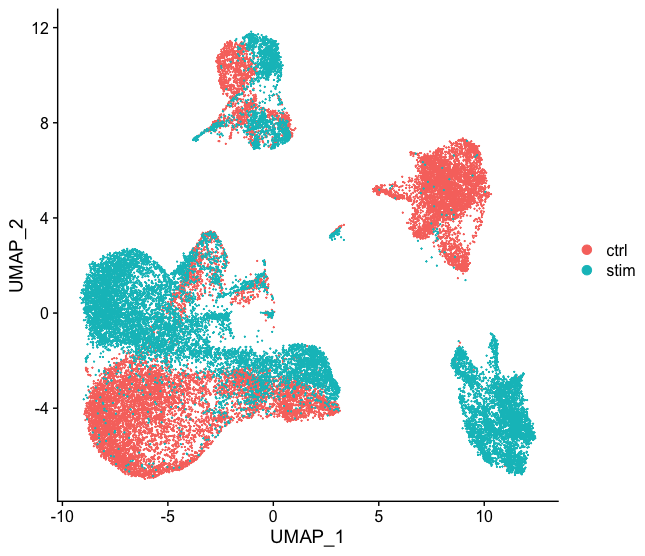
本文主要是翻译,用作参考,还是要看GitHub上的英文原版学习 目标 对于不同条件下的样本,将相同类型的细胞对齐到一起 挑战 对准类似的细胞类型,这样我们就不会因为样品、条件、模式或批次的不...
Aspera批量下载fastq或sra文件(EBI数据库)
本文介绍了如何通过Aspera工具批量下载EBI数据库中的fastq或sra文件。首先,用户需通过conda创建一个新的环境并安装Aspera CLI和sra-tools。在获取到目标文件的下载链接后,可以使用Aspera命令...
单细胞转录组代谢流分析软件COMPASS&MEBOCOST安装
本文详细记录了在Linux系统上安装COMPASS软件的过程,COMPASS是一款用于单细胞转录组代谢流分析的软件。首先,需要创建一个Python 3.9或3.10的conda环境,并确保pandas版本低于2.0.0。接着,安...
生信分析环境搭建–conda+jupyter
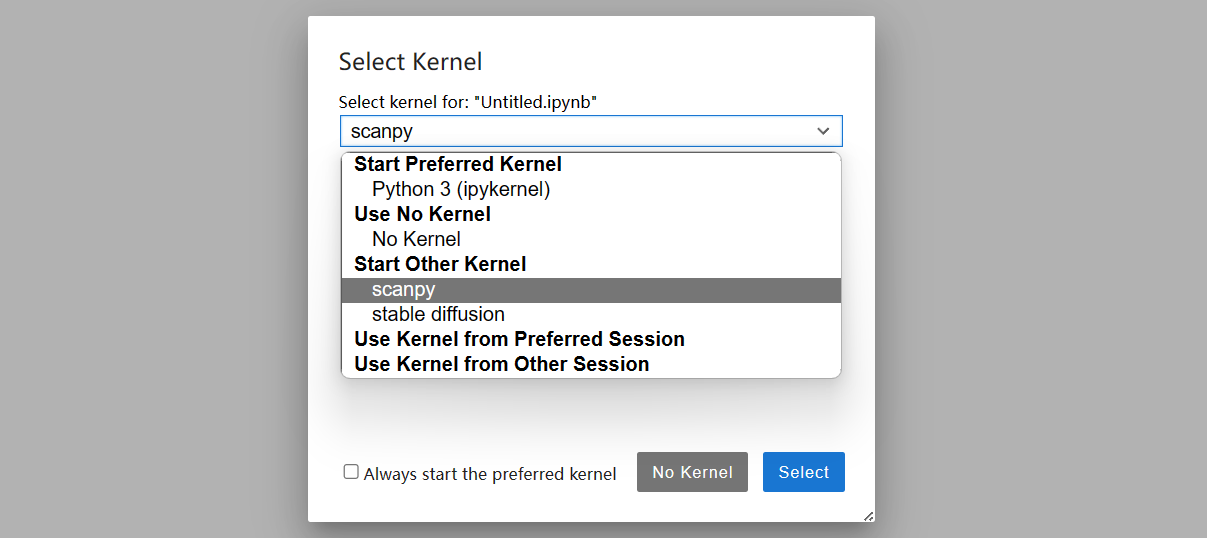
本文介绍了如何使用conda和jupyter notebook搭建生信分析环境,包括安装配置conda、jupyter notebook、scanpy等工具,并演示了在jupyter notebook中调用不同环境的方法。
cellranger count 流程
准备工作:下载参考基因组和数据集 下载参考基因组 # 新建一个ref文件夹存放参考基因组 cd yard mkdir ref cd ref # 下载human GRCH38,大约11G wget https://cf.10xgenomics.com/supp/cell-exp...
cellranger的安装
新建一个文件夹 打开linux终端,运行 mkdir yard 验证一下工作目录 pwd /home/vamond/yard 进入该目录,新建apps文件夹,用于存放cellreanger cd /mnt/home/user.name/yard mkdir apps cd apps ...
使用pheatmap可视化marker基因
在完成单细胞分析基本流程之后,我们获得了各个细胞聚类和相应的marker基因,有多种方式可以可视化marker基因的表达量,seurat包中自带的DoHeatmap()、VlnPlot()以及DotPlot()函数可以很方便的...
使用singleR预测细胞类型
SingleR是用于单细胞RNA测序(scRNAseq)数据的自动注释方法(Aran et al.2019)。给定具有已知标签的样本(单细胞或RNAseq)参考数据集,它将基于与参考数据的相似性标记测试数据集中的新细胞...
对Seurat对象使用harmony方法整合
Harmony方法在2019年发表在上面nature methods, harmony算法与其他整合算法相比的优势: 整合数据的同时对稀有细胞的敏感性依然很好; 省内存; 适合于更复杂的单细胞分析实验设计,可以比较来...