排序
整合-HBC lesson 6.2
本文主要是翻译,用作参考,还是要看GitHub上的英文原版学习 目标 对于不同条件下的样本,将相同类型的细胞对齐到一起 挑战 对准类似的细胞类型,这样我们就不会因为样品、条件、模式或批次的不...
归一化并去除不需要的变异-HBC lesson 6.1
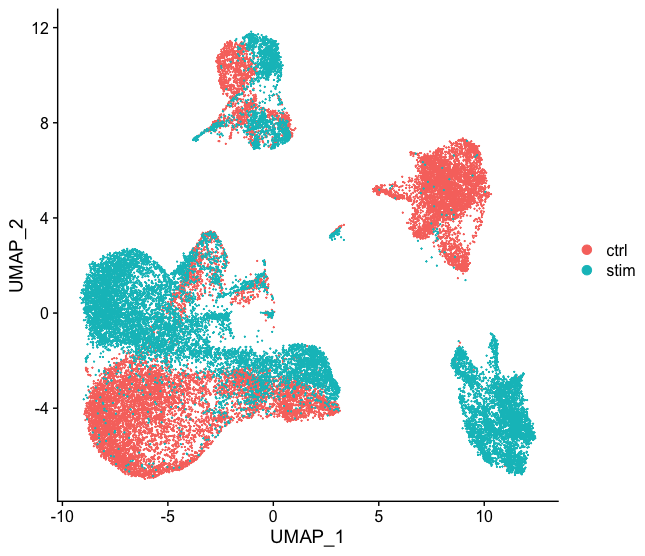
在我们可以聚类细胞并识别不同的潜在细胞类型之前,我们还有几个步骤。我们的数据集有两个样本来自两个不同的条件(控制和刺激),所以整合这些样本,以更好地进行比较可能是有帮助的。我们需要...
归一化与主成分分析-HBC lesson 5
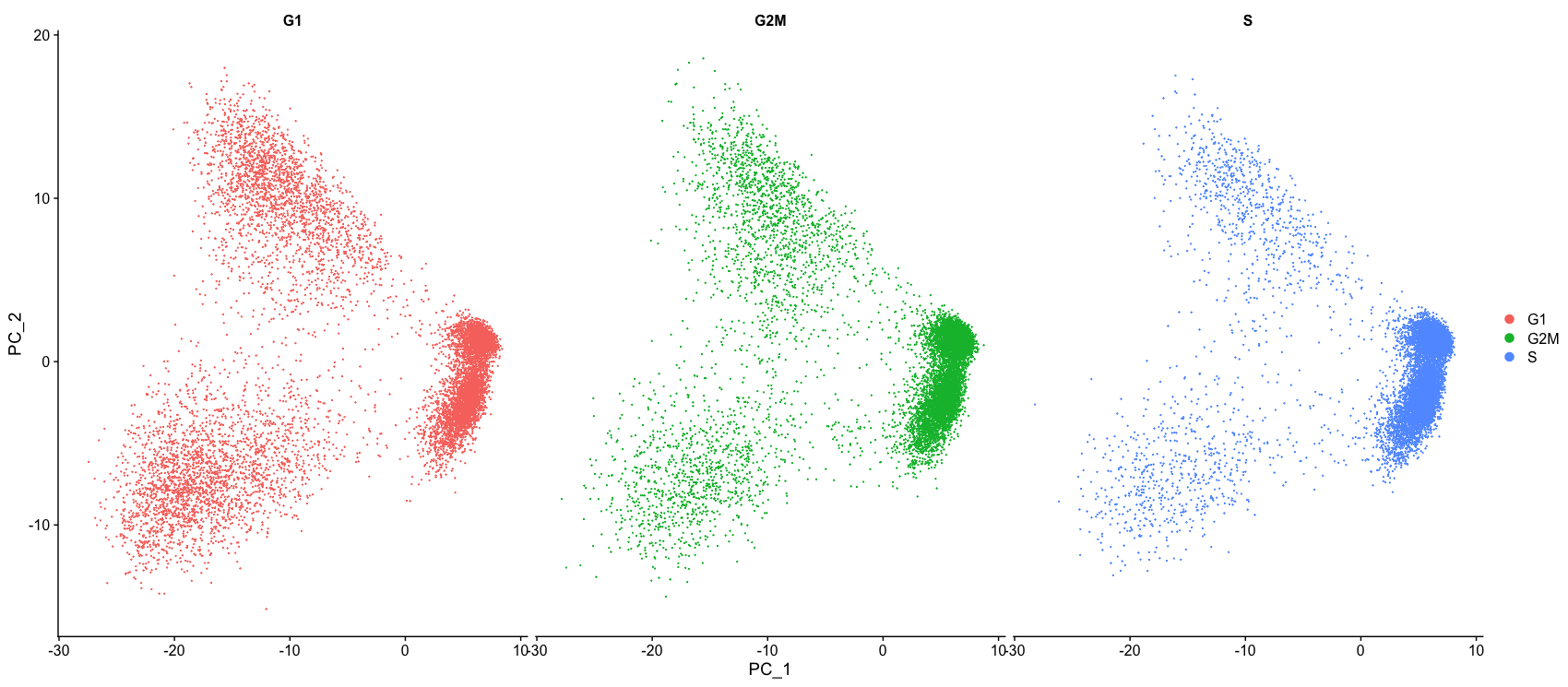
在我们我们获得高质量的单细胞数据后,单细胞RNA测序分析工作流程的下一步是进行聚类。聚类的目标是将不同的细胞类型分离成独特的细胞群。为了执行聚类,我们需要确定细胞间表达差异最大的基因...
Seurat安装
从CRAN安装最新版 4.0+seurat需要R也是4.0+的版本 Seurat is available on CRAN for all platforms. To install, run: # Enter commands in R (or R studio, if installed) install.packages(&#...
QC(质控)-HBC lesson 4
QC(质控)-HBC lesson 4 在这一章你将会学到: 构建质控指标并使用相关图像可视化数据质量 估算质控指标并设置阈值去除低质量的细胞 单细胞数据分析流程的每个步骤都有这自己的目标和挑战。对...
分析准备与读取数据-HBC lesson 3
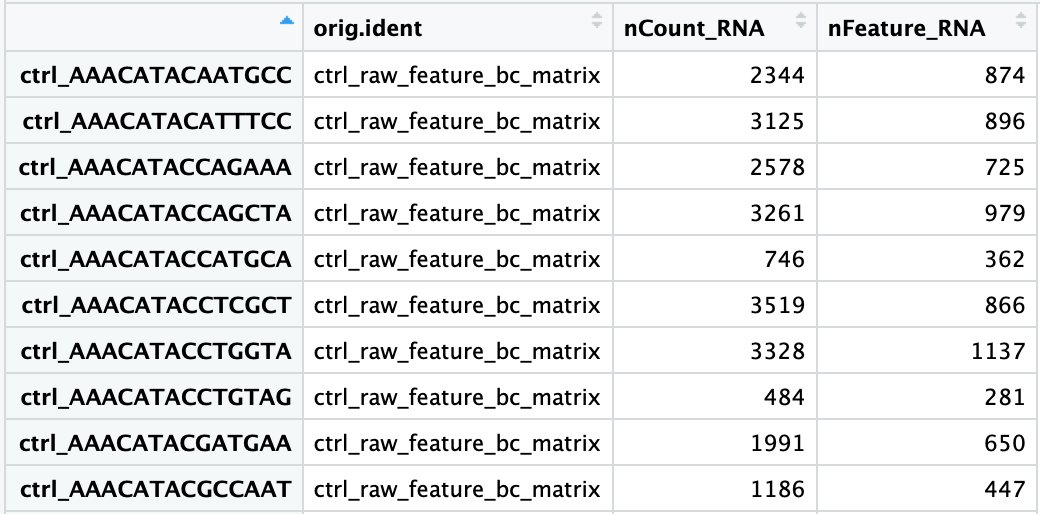
本篇教程将会帮助你理解如何获得单细胞RNA测序实验中的数据。 基因表达定量完成之后,我们需要将这些数据导入R中,以生成可用于执行质控的矩阵。在本课中,我们将讨论计数数据可被导入的格式,...
从原始数据到表达矩阵-HBC lesson 2
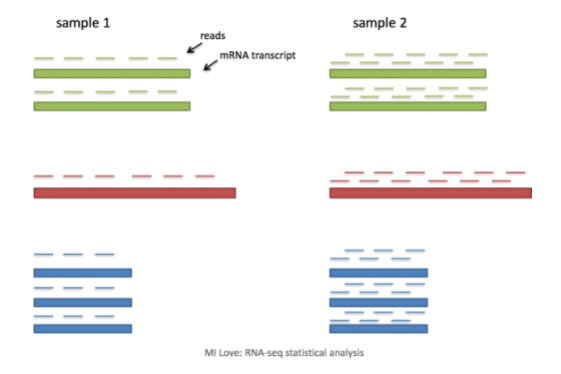
根据所使用的建库方法,单细胞的RNA序列(也称为读取(reads)或标签(tags))将从转录本的3'端(或5'端)(10X Genomics,CEL-seq2,Drop-seq,inDrops)或全长转录本(Smart-seq)获得。 参...
scRNA-seq简介-HBC lesson 1
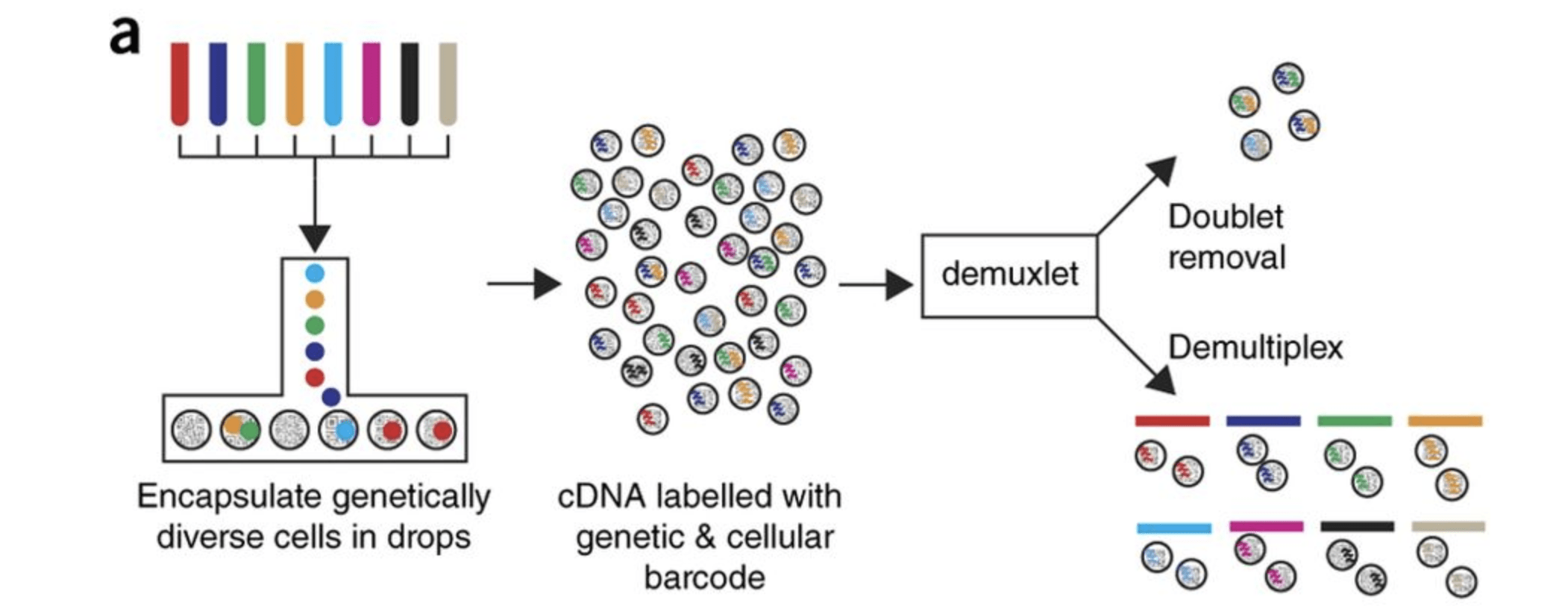
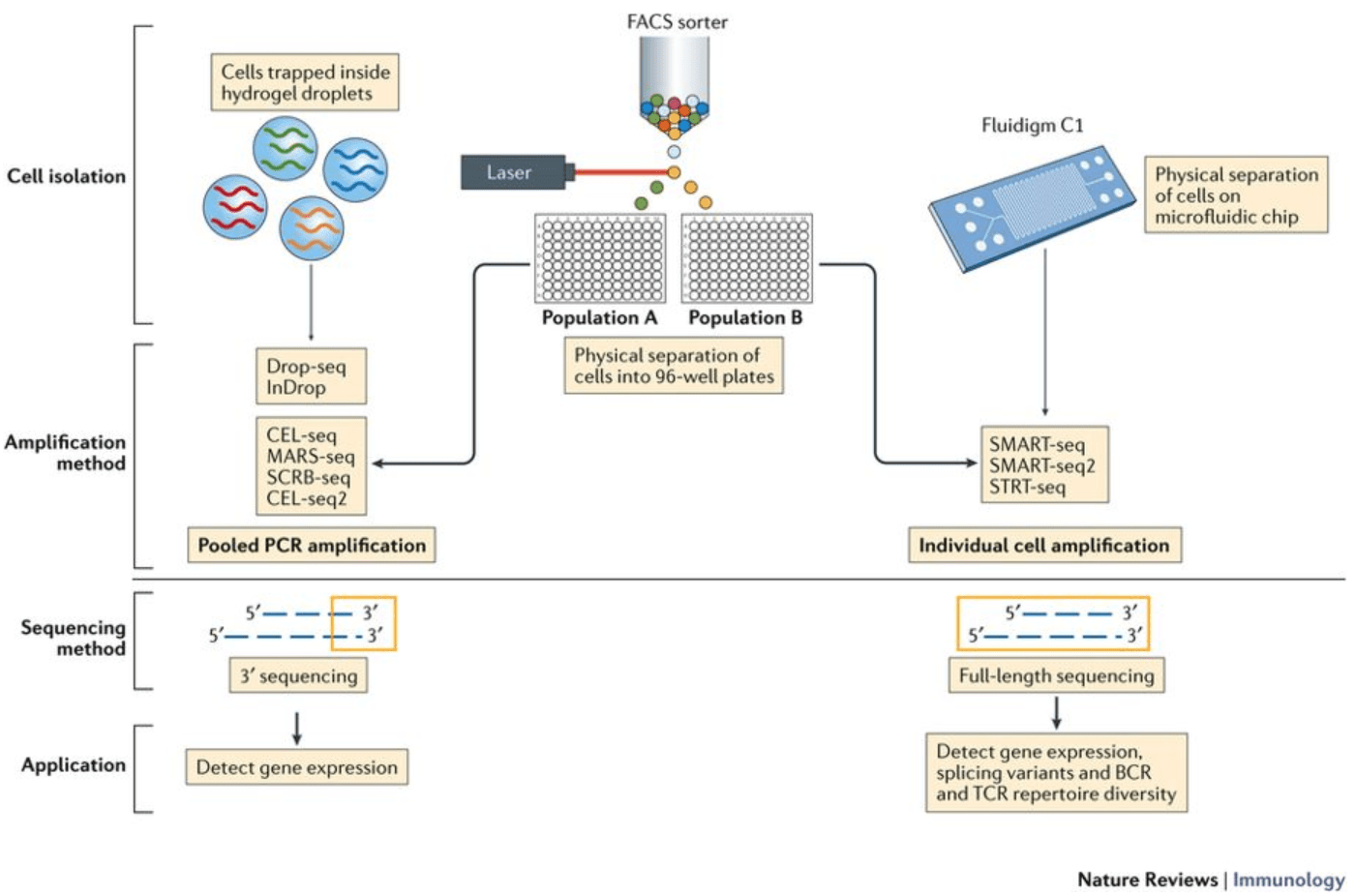
参考链接: https://github.com/hbctraining/scRNA-seq_online/blob/master/lessons/01_intro_to_scRNA-seq.md 我们为什么需要单细胞RNA测序 人类各种组织之间细胞的类型,状态和相互作用差异巨...
“归一化”与“标准化”
参考链接: https://mp.weixin.qq.com/s/6ioR3JE0wKg6M-YAsLBcTA 关于归一化和标准化的翻译看了很多中文资料,发现还是有争议的,在seurat中主要是两个函数:NormalizeData()和ScaleData() ,其...
单细胞聚类resolution参数的选择
在进行细胞聚类时有两个参数的选择对下游分析的结果影响很大 一个是纳入分析的PCs数,也就是dims参数,选择方法见 关于“数据的维度”(dims参数)的选择 ; 另一个是resolution参数,官方推荐根...