安装omicverse docker版
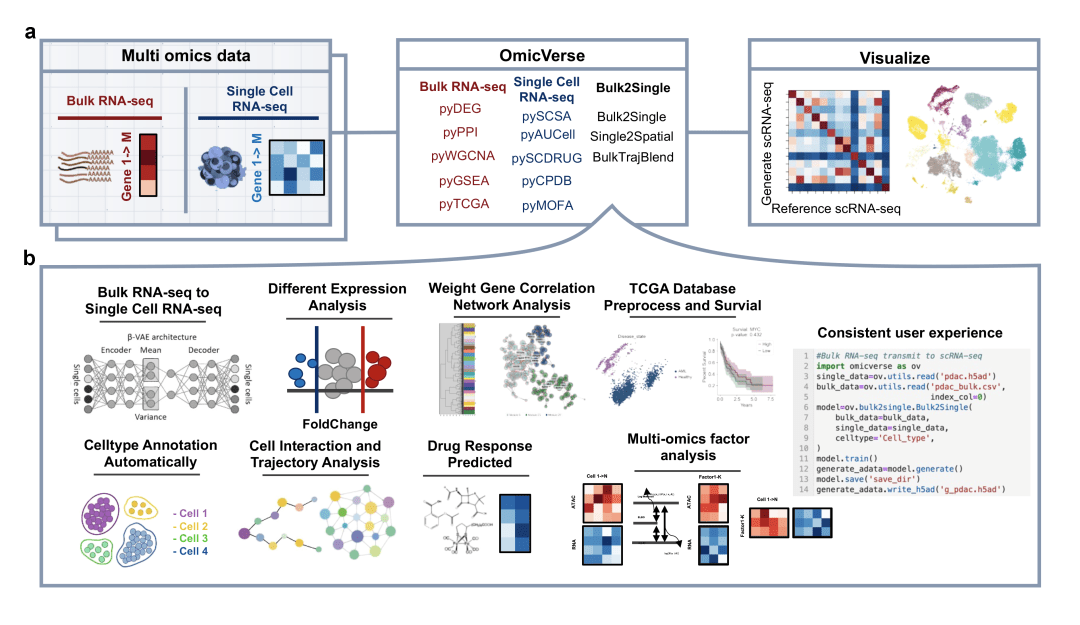
Omicverse简介
与R相比,python在处理大型数据时具有快速高效,内存占用优秀,可以便捷调用GPU加速等特点。Omicverse构建了一套基于python的高通量测序数据的分析框架,其中囊括了bulk rna seq,single cell等分析要用的大部分主流软件。以下是作者的自述:
omicverse是在我大三保研做Bulk RNA-seq时提出的,当时为了自己的方便,就把差异表达分析(DEG),加权基因共表达分析(WGCNA),通路富集分析(GSEA)等整合进了一个叫Pyomic的包中方便自己调用,当时的野心很大,想把整个组学分析都用Python来完成 (笑) 。
现在博士一年级,也做了快两年的单细胞分析,中途用了很多包,但不同的包之间,对于格式的需求往往是不一样的,而且有的包本身并没有好好利用GPU的计算优势,并且大部分的包所作的图与CNS上的图差别还是比较大的。所以我就把当时的Pyomic给捡了起来,把我做分析用到的所有包都塞进了omicverse里面,统一了它们的输入输出,并对部分可以优化的算法进行了优化,以及给出了一些新的可视化函数来帮助研究人员呈现结果。可能你会想,这不就是一个整合的包吗?但其实不是。
我通过大量的基准测试,以及相关的paper,整理出了一套最适的Python单细胞分析流程,Bulk RNA-seq分析流程与可视化。
我提出了一种新的生物学算法 “BulkTrajBlend”,解决了单细胞测序中由于通量限制导致的细胞不连续现象。
·

记录安装失败的过程
使用conda或者pip安装omicverse尝试了好久依然无法解决依赖问题,非常难装,没错即使是conda也不行,后来看到官方有提供docker部署的方式,做了以下尝试:
由于我想安装gpu版本,我的cuda是12.1,所以选择以下命令安装:
#create a folder to connect the docker and host
mkdir '/path/analysis'
#path is the folder where you store your data locally
local_data_path='/path/analysis'
#cuda121
docker run -it -v "$local_data_path":/analysis -p 8899:8899 --gpus all starlitnightly/omicverse:py310_cu121_latest
但是我发现docker内无法调用gpu导致无法启动:
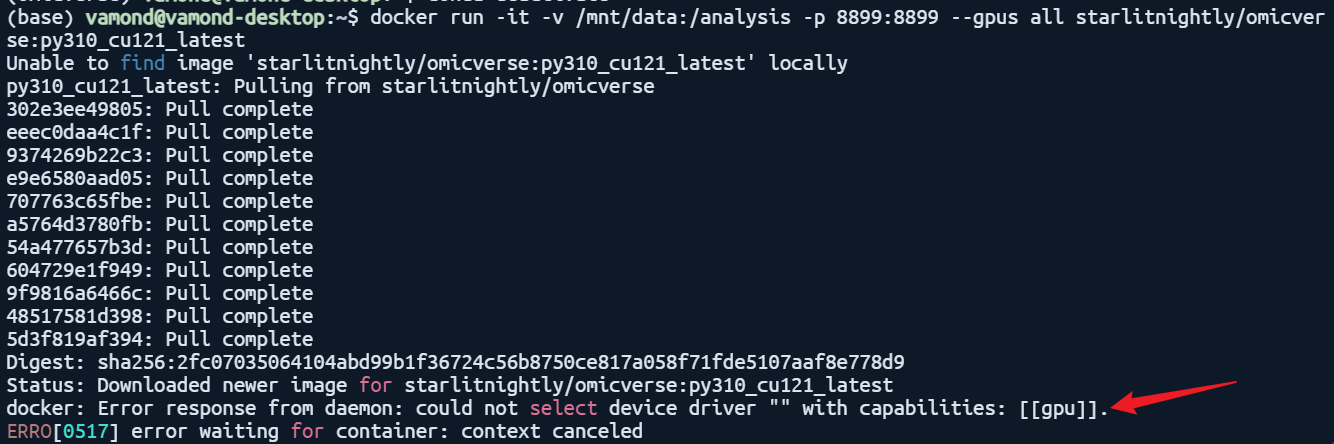
从头再来
查询后得知原来是需要先安装nividia container toolkit,按照英伟达教程安装:
安装NVIDIA Container Toolkit
curl -fsSL https://nvidia.github.io/libnvidia-container/gpgkey | sudo gpg --dearmor -o /usr/share/keyrings/nvidia-container-toolkit-keyring.gpg \
&& curl -s -L https://nvidia.github.io/libnvidia-container/stable/deb/nvidia-container-toolkit.list | \
sed 's#deb https://#deb [signed-by=/usr/share/keyrings/nvidia-container-toolkit-keyring.gpg] https://#g' | \
sudo tee /etc/apt/sources.list.d/nvidia-container-toolkit.list
sudo apt-get update
sudo apt-get install -y nvidia-container-toolkit
配置NVIDIA Container Toolkit
在此之前需要确定已经安装好了docker并能正常运行,NVIDIA Container Toolkit也通过上述代码安装完成没有报错。下面配置:
sudo nvidia-ctk runtime configure --runtime=docker
重启docker:
sudo systemctl restart docker
运行omicverse docker
docker run -it -v /mnt/data:/analysis -p 8899:8899 --gpus all starlitnightly/omicverse:py310_cu121_latest
上面/mnt/data是我的分析目录,请自行替换。
然后会自动安装需要的包,这个应该是在docker容器内安装的:
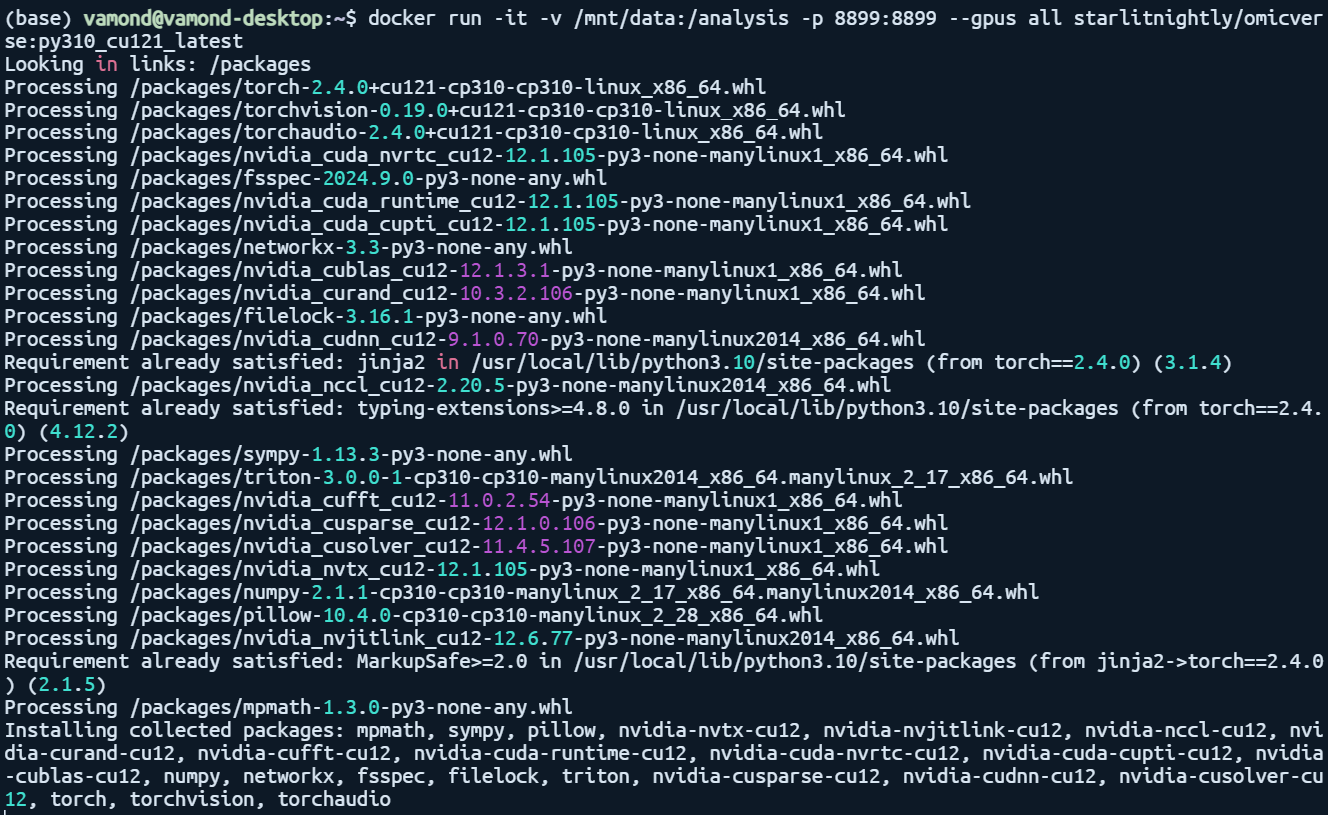
当终端出现以下输出,即表明安装完成,jupyter lab已经启动:
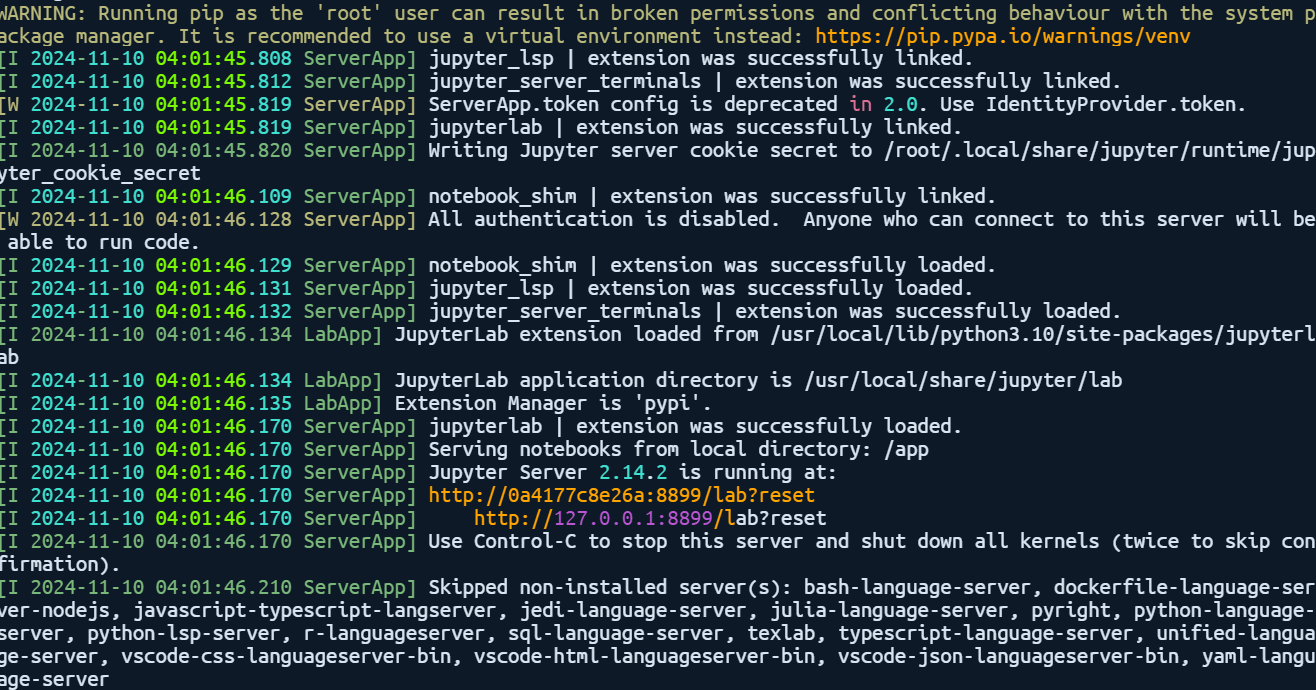
检查
安装完成之后通过ip:端口的方式访问即可进入jupyter lab页面,尝试一下import,没有报错表明成功

但是我们可以看到官方docker内有没有设置好默认目录,官方的命令是把我们的自定义目录映射到了容器内的/analysis,但是我发现这个jupyter lab的默认工作目录似乎是在/app目录,那么我们需要修改一下命令:
首先在终端里ctrl+C将目前的jupyter关闭,然后使用以下命令重新启动。
注意,此命令会重新创建容器,意味着会自动重新安装所有依赖,时间会比较久哦。
docker run -it --gpus all -v /mnt/data:/app -p 8899:8899 starlitnightly/omicverse:py310_cu121_latest
此时再看jupyter lab页面,已经出现了我自己的文件了
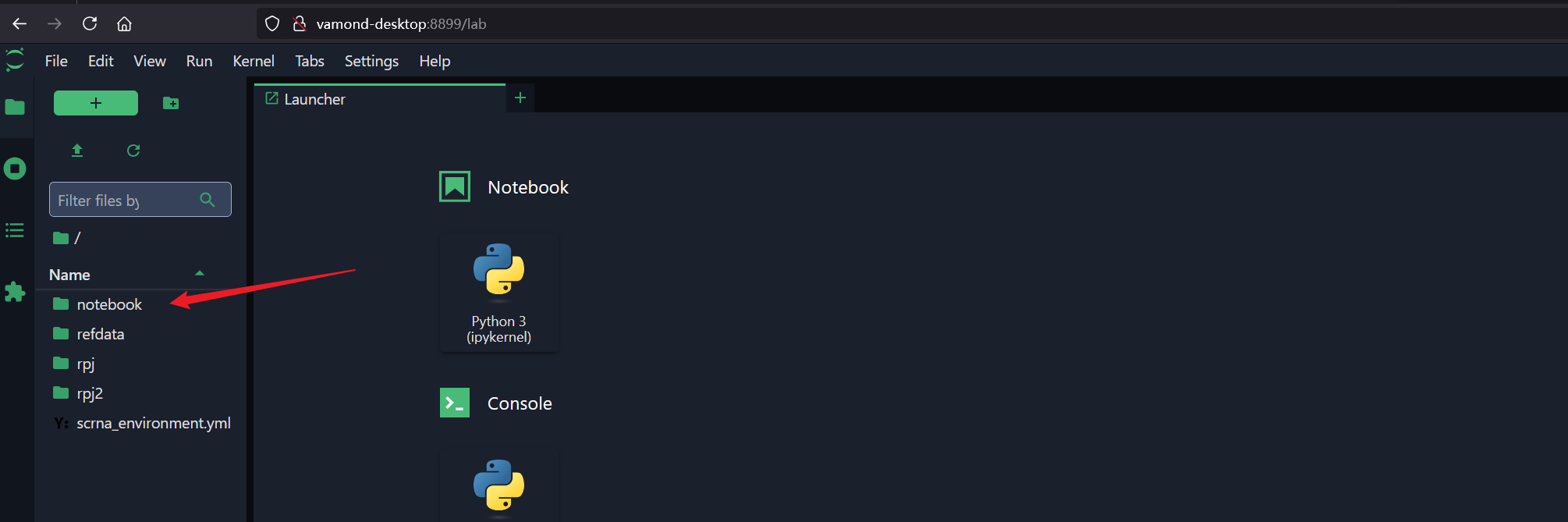
容器管理
后续如果关闭了容器,但是容器本身并没有被删除,可以直接启动之前的容器即可,不需要使用 docker run 命令创建一个新的容器。参考以下命令:
# 列出所有容器(包括未运行的)
docker ps -a
我这里输出如下
(base) vamond@vamond-desktop:~$ docker ps -a
CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES
4054dac790f7 starlitnightly/omicverse:py310_cu121_latest "sh -c 'pip install …" 5 weeks ago Exited (137) 4 weeks ago infallible_cartwright
这时候,我们可以复制容器编号,我这里示例为4054dac790f7
# 启动指定容器
docker start 4054dac790f7
这样做的好处是,我们不需要每一次开机都用 docker run 命令创建一个新的容器;此外,如果我们在容器内安装过其他软件,在重新启动容器之后也不需要重新安装。
如果了解 docker 的话,我们当然还可以基于 omicverse 的官方镜像进一步构建自己的镜像,可以包含一些原始镜像没有的软件或设置。有兴趣的可以自行检索 docker 的使用教程。
常见问题
- 如果你不需要调用gpu加速,直接使用cpu版本的docker即可,大概率不需要这么复杂。
- 在拉取镜像之前请自行安装好docker,如果没有科学上网还需要配置一个可用的源(加速url),才能正常拉取镜像,下次有时间再详细写一下docker怎么用。
- 初次运行docker run时会自动拉取镜像,大约5Gb的数据量,需要保证网络没问题,否则会运行失败,建议使用
- 启动容器命令的文件夹映射和端口映射是可以根据自己需要调整的,不一定跟官方一致。比如你启动jupyter用的是默认的8888端口,那么端口映射可以写
8888:8888,如果系统有防火墙记得开放端口才能访问。 - 最后解决映射目录的问题,实际上进入容器内修改jupyter工作目录也是可以实现的,但是最简单的方法应该还是直接改一下命令。
-
docker run之后终端不能关闭,如果需要后台运行,可以使用nohup命令
参考
- Omicverse概述
- Dockerhub-omicverse
- NVIDIA Container Toolkit安装教程
- omicverse使用教程
- 杏仁AI智能对话解决一些问题
本文介绍了如何在Linux系统上安装Omicverse的Docker版本。Omicverse是一个基于Python的高通量测序数据分析框架,具有统一输入输出和改进的可视化功能。作者首先尝试通过conda和pip方式安装,但遇到依赖问题难以解决,最终选择使用Docker安装。为此,需要先安装NVIDIA Container Toolkit以支持GPU。文中详细讲解了安装步骤,包括添加NVIDIA软件源、安装容器工具包、配置容器运行时并重启Docker服务。成功部署后,通过Docker命令启动Omicverse,并映射本地数据目录,以便在Jupyter Lab中进行生物信息学分析。











暂无评论内容