因为课题研究需要结合参考一篇已发表的单细胞数据,由于该文章只提供的原始fastq文件,所有要下载下来从上游分析开始做起。(如果是是做数据库生信挖掘的也需要下载文件哦)一开始尝试的使用http和ftp方式下载,发现速度很慢,而且很容易下载到一半就断开连接了,最过分的是竟然不能断点重连继续下载。好不容易显示下载完了,进行md5校验发现竟然不对! 于是在网上找了一些资料,发现可以通过aspera进行高速下载,学习后记录于此。
IBM aspera 官网
在数据库中定位测序文件
文章中提到数据提交到了NCBI bioproject 数据库,编号PRJNA562645,于是可以通过这个信息在ncbi bioproject中检索即可,现在ncbi的测序数据都是用sra文件保存的,据说可以节省空间,提高传输效率,但是考虑到sra文件下载之后还需要进行转换为fastq才能进行后续处理,并且ncbi似乎没有提供文件的hash码,担心万一网络有问题丢包什么的也不确定文件是否完整。其实ncbi也提供fastq文件,但是个人认为不是很好找,于是找到另一个数据库European Nucleotide Achive (EBI),可以直接下载fastq文件,并且提供的了md5码。
EBI数据库官网
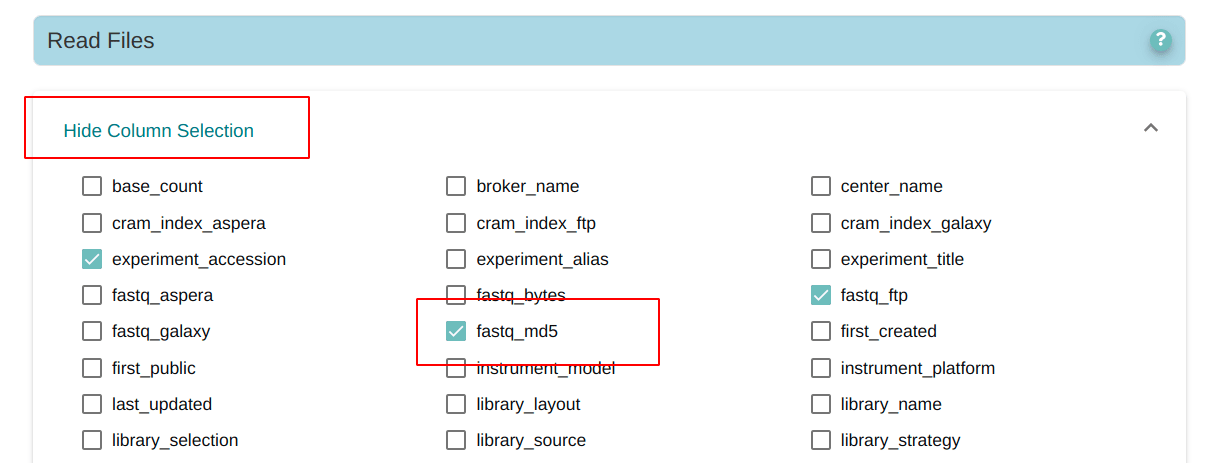
在EBI中检索到我们需要的项目文件后,记得要在这里选择一下column,可以让他把md5码显示出来,便于我们后面下载完以后进行文件校验。
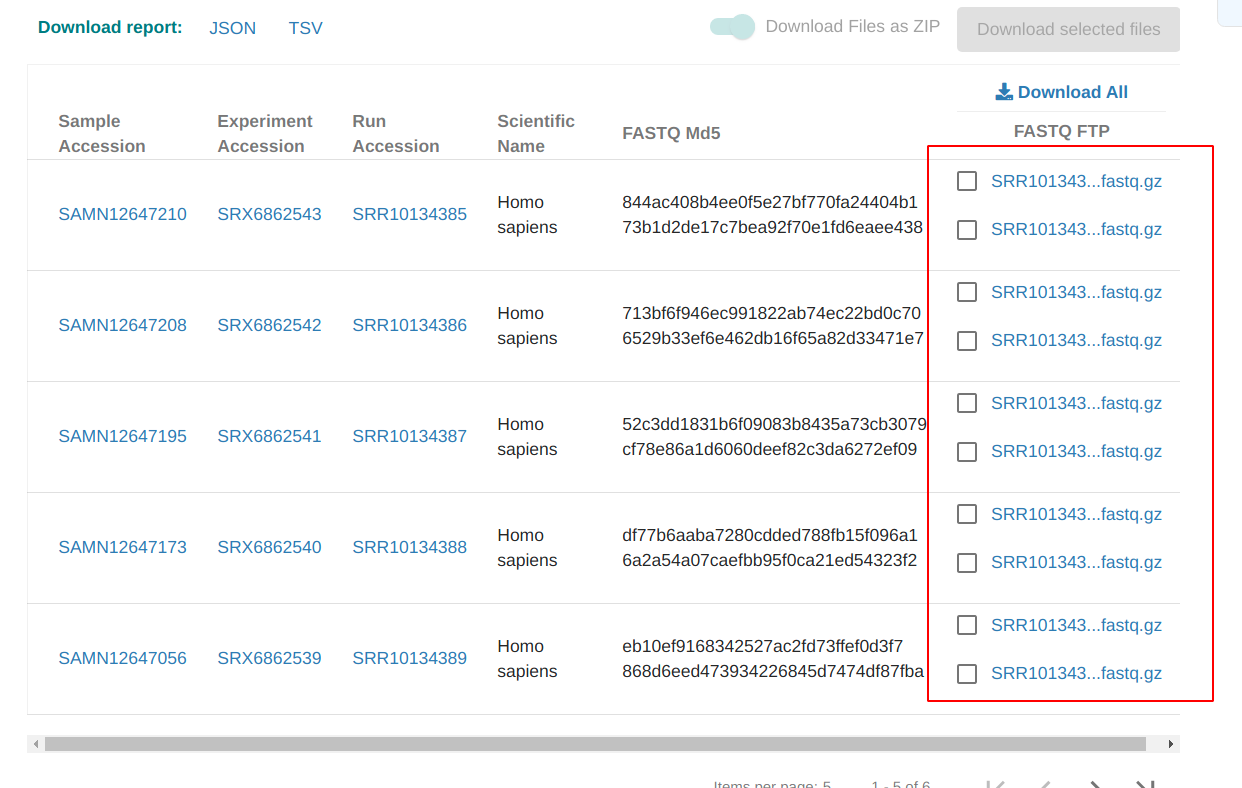
然后FASTQ FTP一列就是我需要下载的文件了:
右键复制下载链接
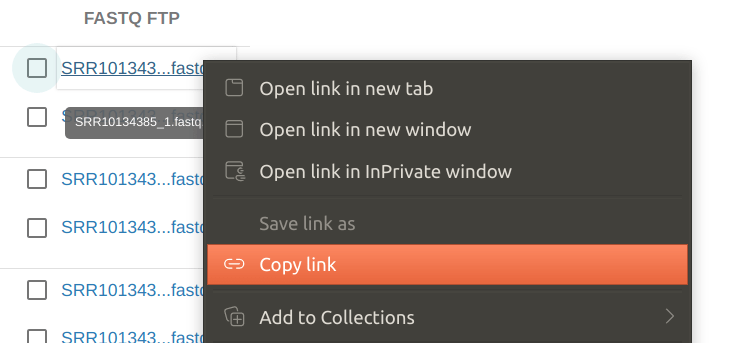
先观察一下下载链接是啥样的,默认是ftp协议的下载方式:
ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR101/085/SRR10134385/SRR10134385_1.fastq.gz如果是勾选了多个文件,网站会生成一个zip压缩文件,通过https协议下载,这种方法不推荐。
其实如果是普通的转录组文件的话,可以通过linux终端中的curl或者wget命令进行下载,但是单细胞的数据通常都得几十个G,就需要用到开头提到的aspera进行高速稳定的下载。
Aspera的安装与使用

2023年3月更新:
使用conda安装又快又好,下文的安装方法已不再推荐。conda命令如下,不再解释,详见生信技能树推文。
conda create -n downloadconda activate downloadconda install -y -c hcc aspera-cliconda install -y -c bioconda sra-toolswhich ascp## 一定要搞清楚你的软件被conda安装在哪ls -lh ~/miniconda3/etc/asperaweb_id_dsa.openssh
安装
PS:我是在自己的电脑(unbuntu 18.04)上直接安装的,如果是用的服务器的话,先ssh登录一下即可。 我安装的是3.10.0版本的aspera connect:
# 下载
wget https://ak-delivery04-mul.dhe.ibm.com/sar/CMA/OSA/092u0/0/ibm-aspera-connect-3.10.0.180973-linux-g2.12-64.tar.gz
# 解压
tar -xvf ibm-aspera-connect-3.10.0.180973-linux-g2.12-64.tar.gz
# 解压后得到一个脚本文件,运行该脚本,即可完成自动安装
sh ibm-aspera-connect-3.10.0.180973-linux-g2.12-64.sh
# 所有安装文件都在~/.aspera/connect目录下,添加环境变量
echo 'export PATH=~/.aspera/connect/bin/:$PATH' >> ~/.bashrc
# 使环境变量生效
source ~/.bashrc
# 查看ascp可执行文件所在的路径,应该是:~/.aspera/connect/bin/ascp
which ascp
# 看程序是否能正常运行
ascp -h上面在运行sh脚本的那一步,我在实际操作的过程中遇到小问题,无法执行,报错Syntax error: "(" unexpected,最后解决方案是参考以下博客:
linux shell脚本报错:Syntax error: “(” unexpected_兰兰的博客-CSDN博客
是因为.sh文件运行时,重定向为dash启动导致的,终端运行以下代码即解决:
sudo dpkg-reconfigure dash或者在sh脚本第一行加入#!/bin/bash
使用
本文只记录了我下载过程的实际操作,也就是下载ebi数据库中的fastq文件的方法,其他数据库及参数可以参考下方链接:
Aspera:基因组数据高速下载利器,以NCBI和EBI数据下载为例
单个文件的下载
运行ascp命令,ascp —help 查看帮助文档。
ascp -QT -l 300m -P33001 -k1 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR101/090/SRR10134390/SRR10134390_1.fastq.gz .主要参数说明:
- -T 免密传输
- -l 最大传输速度,后面300m,表示尝试以300M/s的速度进行传输,亲测基本上可以跑满带宽,校园有线网络下达到了95M/s ,而且基本很稳定。
- -P 文档里说是
Preserve file timestamp,也有人说是端口,后面一般用33001,如果不加这个参数会报错,填别的数字也会报错。 - -k 断点续传功能,一般设置为1表示打开。如果真的万一下载到一般出现网络问题了,这个参数就是神器了,不需要删除下载了一半的文件再从头开始下载,节省时间。
- -i 免密从NCBI或EBI下载的私钥,安装完成就有,位于
~/.aspera/connect/etc/asperaweb_id_dsa.openssh(这个aspera是IBM开发的商业软件,但是ncibi和ebi都是他的客户,我们实际上是薅 ncbi/ebi 的羊毛才能够免费使用的。) era-fasp@fasp.sra.ebi.ac.uk就是用户和服务器地址了,把上面复制得到的ftp文件url中ftp://ftp.sra.ebi.ac.uk改掉,后面文件路径保持- 最后一个
.是表明下载下来以后文件保存在当前路径中,也可以指定为其他路径
多个文件的批量下载
# 新建一个文档写入文件列表
touch file.lst
# 编辑
gedit file.lst比如本示例中的文件列表是这么写的,共有12个文件:
/vol1/fastq/SRR101/085/SRR10134385/SRR10134385_1.fastq.gz
/vol1/fastq/SRR101/085/SRR10134385/SRR10134385_2.fastq.gz
/vol1/fastq/SRR101/086/SRR10134386/SRR10134386_1.fastq.gz
/vol1/fastq/SRR101/086/SRR10134386/SRR10134386_2.fastq.gz
/vol1/fastq/SRR101/087/SRR10134387/SRR10134387_1.fastq.gz
/vol1/fastq/SRR101/087/SRR10134387/SRR10134387_2.fastq.gz
/vol1/fastq/SRR101/088/SRR10134388/SRR10134388_1.fastq.gz
/vol1/fastq/SRR101/088/SRR10134388/SRR10134388_2.fastq.gz
/vol1/fastq/SRR101/089/SRR10134389/SRR10134389_1.fastq.gz
/vol1/fastq/SRR101/089/SRR10134389/SRR10134389_2.fastq.gz
/vol1/fastq/SRR101/090/SRR10134390/SRR10134390_1.fastq.gz
/vol1/fastq/SRR101/090/SRR10134390/SRR10134390_2.fastq.gz保存之后运行以下命令,进行批量下载:
ascp -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -P33001 -l 300M -T -k1 --mode recv --host fasp.sra.ebi.ac.uk --user era-fasp --file-list ./file.lst .注意批量下载这里,用户和服务器地址是分开写的两个参数哦。
--host fasp.sra.ebi.ac.uk (将原下载链接中的 ftp://ftp.sra.ebi.ac.uk 改称这个)
--user era-fasp (也就是@前面的那个部分)
批量下载这里还多了一个mode参数,写的是 recv 表示接收文件,该参数不可省略。
md5校验
通过aspera下载的文件一般情况下不会出什么问题,但是为了保险起见,本着严谨的科研作风,我们还是来校验一下文件。
由于文件比较大,在本地使用命令行计算md5可能会比较麻烦,发现了一个很好的网页工具,使用前端计算hash值,并且内存占用很低。

完成后将这个md5码和EBI提供的md5比对一下即可,一致的则表示下载的文件完整。
由于文件比较大,这一步也是比较耗费时间的,甚至比下载还慢,不过可能跟硬盘的读写速度有关。











暂无评论内容